Integrase
| Dominio de unión ao Zn de integrase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Estrutura en solución do dominio de unión ao Zn N-terminal da integrase do VIH-1 (forma e), RMN, 38 estruturas | |||||||||
| Identificadores | |||||||||
| Símbolo | Integrase_Zn | ||||||||
| Pfam | PF02022 | ||||||||
| InterPro | IPR003308 | ||||||||
| SCOPe | 1wjb / SUPFAM | ||||||||
| |||||||||
| Dominio central de integrase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
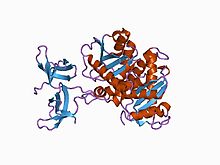 Estrutura cristalina da integrase de dous dominios do virus do sarcoma de Rous. | |||||||||
| Identificadores | |||||||||
| Símbolo | rve | ||||||||
| Pfam | PF00665 | ||||||||
| Pfam clan | CL0219 | ||||||||
| InterPro | IPR001584 | ||||||||
| SCOPe | 2itg / SUPFAM | ||||||||
| |||||||||
| Dominio de unión ao ADN de integrase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Estrutura cristalina da integrase de dous dominios do virus do sarcoma de Rous. | |||||||||
| Identificadores | |||||||||
| Símbolo | IN_DBD_C | ||||||||
| Pfam | PF00552 | ||||||||
| InterPro | IPR001037 | ||||||||
| SCOPe | 1ihw / SUPFAM | ||||||||
| |||||||||
Unha integrase retroviral (IN), frecuentemente denominada integrase, é un encima producido por un retrovirus (como o VIH) que integra (formando enlaces covalentes) a súa información xenética na da célula hóspede que infecta.[1] As INs retrovirais non se deben confundir coas integrases de fagos (recombinases) usadas en biotecnoloxía, como a integrase do fago λ utilizada na recombinación específica de sitio. Por outra parte, os retrotransposóns con LTR conteñen unha polimerase con varias actividades, entre elas a de integrase.
O complexo macromolecular dunha macromolécula de IN unida aos extemos do ADN viral foi denominado intasoma; a IN é un compoñente clave deste complexo e do complexo de preintegración retroviral.[2]
Estrutura
[editar | editar a fonte]Todas as proteínas IN retrovirais conteñen tres dominios canónicos, conectados por enlazadores (linkers) flexibles:[3][4]
- un dominio de unión ao Zn HH-CC N-terminal (un feixe de tres hélices estabilizado por coordinación dun catión Zn(II)),
- un dominio central catalítico (pregamento RNaseH),
- un dominio de unión ao ADN C-terminal (pregamento SH3).
Obtivéronse as estruturas cristalinas e de RMN de cada dominio por separado e de construtos de dous dominios das integrases do VIH-1, VIH-2, virus da inmunodeficiencia simia, e virus do sarcoma de Rous, e as primeiras desas estruturas determináronse en 1994.[5][6] Os datos bioquímicos e estruturais suxiren que a IN retroviral funciona como un tetrámero (dímero de dímeros), e os seus tres dominios son importantes para a multimerización e a unión ao ADN viral.[7] Ademais, varias proteínas do hóspede celular interaccionan coa IN para facilitar o proceso de integración: por exemplo, o factor do hóspede proteína asociada á cromatina humana LEDGF, únese estreitamente á integrase do VIH e dirixe o complexo de preintegración do VIH cara a xenes altamente expresados para a súa integración.[8]
O virus escumoso humano (HFV), un axente inocuo para os humanos, ten unha integrase similar á do VIH e por iso serve como modelo da función da IN do VIH; determinouse unha estrutura cristalina da integrase do HFV ensamblada aos extremos do ADN viral.[6]
Función e mecanismo
[editar | editar a fonte]A integación ocorre despois da produción do ADN viral liñal bicatenario pola ADN polimerase dependente de ARN/ADN viral chamada transcritase inversa.[9]
A principal función da IN é inserir o ADN viral no ADN cromosómico do hóspede, un paso esencial para a replicación do VIH. A integración é un "punto de non retorno" para a célula, que se converte nun transportador permanente do xenoma viral (provirus). A integración é en parte responsable da persistencia das infeccións retovirais.[10] Despois da integración, pode ter lugar a expresión dos xenes virais e a produción de partículas de novos virións inmediatamente ou nalgún momento do futuro, a cronoloxía depende da actividae do locus cromosómico que hospeda o provirus.[4]
As INs retrovirais catalizan dúas reaccións:[4]
- Procesamento 3', no cal se eliminan dous ou tres nucleótidos dun ou de ambos os extremos 3' do ADN viral para expoñer un dinucleótido CA invariante.
- Reacción de transferencia da febra, na cal os extremos 3' procesados do ADN viral son enlazados covalentemente ao ADN cromosómico do hóspede.
Ambas as reaccións son catalizadas no mesmo sitio activo, e implican a transesterificación sen implicar un intermediario covalente proteína-ADN (a diferenza das reaccións catalizadas pola Ser/Tyr recombinase).[4]
No VIH
[editar | editar a fonte]
A integrase do VIH é unha proteína viral de 32 kDa que consta de tres dominios: N-terminal, dominio central catalítico, e C-terminal, cada un dos cales ten distintas propiedades e funcións que contribúen á eficacia da integrase do VIH.[3]
O N-terminal está composto por 50 residuos de aminoácidos que conteñen unha secuencia que quela ións cinc, aumentando máis a actividade encimática do dominio central catalítico.[3] Como a quelación de metais é vital na eficacia da integrase, é unha diana para o desenvolvemento de terapias retovirais.[3]
O dominio central catalítico, igual que o N-terminal, contén residuos de aminoácidos moi conservados -Asp64, Asp116, Glu152-, xa que o motivo DDE (Asp-Asp-Glu) conservado contribúe ás funcións de endonuclease e polinucleótido transferase da integrase. As mutacións nestas rexións inactivan a integrase e impiden a integración no xenoma.[3]
O dominio C-terminal únese ao ADN do hóspede de forma non específica e estabiliza o complexo de integración.[3]
Mecanismo de integración
[editar | editar a fonte]Despois da síntese do xenoma de ADN bicatenario do VIH, a integrase únese ao longo tramo de repeticións en tándem que flanquean o xenoma en ambos os extremos. Usando a súa actividade endonucleolítica, a integrase corta un di- ou trinucleótido en ambos os extremos 3' do xenoma nun procesamento coñecido como procesamento 3'.[11] A especificidade de corte mellora co uso de cofactores como o Mn2+ e o Mg2+, que interaccionan co motivo DDE do dominio central catalítico, que actúan como cofactores para a función de integrase.[11]
Os grupos 3'OH que se xeran distorsionan os enlaces fosfodiéster do ADN hóspede por medio dun ataque nucleofílico de tipo SN2.[6] Os extremos 3' están enlazados covalentemente ao ADN diana. Os tramos que sobresaen no extremo 5' do xenoma viral son despois cortados usando os encimas de reparación do hóspede, eses mesmos encimas crese que son responsables da integración do extremo 5' no xenoma do hóspede formando o provirus.[6][11]
Terapia antirretroviral
[editar | editar a fonte]En novembro de 2005, os datos dun estudo en fase II dun inhibidor da integrase do VIH en investigación, MK-0518, demostraban que o composto tiña unha potente actividade antiviral. En outubro de 2007, a FDA dos Estasdos Unidos aprobou o inhibidor da integrase Raltegravir (MK-0518, nome comercial Isentress). Un segundo inhibidor da integrase, elvitegravir, foi aprobado nos Estados Unidos en agosto de 2012.
Notas
[editar | editar a fonte]- ↑ Beck BJ, Freudenreich O, Worth JL (2010). "Patients with Human Immunodeficiency Virus Infection and Acquired Immunodeficiency Syndrome". Massachusetts General Hospital Handbook of General Hospital Psychiatry. Elsevier. pp. 353–370. ISBN 9781437719277. doi:10.1016/b978-1-4377-1927-7.00026-1.
- ↑ Masuda T (2011). "Non-Enzymatic Functions of Retroviral Integrase: The Next Target for Novel Anti-HIV Drug Development". Frontiers in Microbiology 2: 210. PMC 3192317. PMID 22016749. doi:10.3389/fmicb.2011.00210.
- ↑ 3,0 3,1 3,2 3,3 3,4 3,5 Jóźwik IK, Passos DO, Lyumkis D (setembro de 2020). "Structural Biology of HIV Integrase Strand Transfer Inhibitors". Trends in Pharmacological Sciences 41 (9): 611–626. PMC 7429322. PMID 32624197. doi:10.1016/j.tips.2020.06.003.
- ↑ 4,0 4,1 4,2 4,3 Delelis O, Carayon K, Saïb A, Deprez E, Mouscadet JF (decembro de 2008). "Integrase and integration: biochemical activities of HIV-1 integrase". Retrovirology 5 (1): 114. PMC 2615046. PMID 19091057. doi:10.1186/1742-4690-5-114.
- ↑ Lodi PJ, Ernst JA, Kuszewski J, Hickman AB, Engelman A, Craigie R, et al. (agosto de 1995). "Solution structure of the DNA binding domain of HIV-1 integrase". Biochemistry 34 (31): 9826–9833. PMID 7632683. doi:10.1021/bi00031a002.
- ↑ 6,0 6,1 6,2 6,3 Choi E, Mallareddy JR, Lu D, Kolluru S (outubro de 2018). "Recent advances in the discovery of small-molecule inhibitors of HIV-1 integrase". Future Science OA 4 (9): FSO338. PMC 6222271. PMID 30416746. doi:10.4155/fsoa-2018-0060.
- ↑ Hare S, Di Nunzio F, Labeja A, Wang J, Engelman A, Cherepanov P (xullo de 2009). Luban J, ed. "Structural basis for functional tetramerization of lentiviral integrase". PLOS Pathogens 5 (7): e1000515. PMC 2705190. PMID 19609359. doi:10.1371/journal.ppat.1000515.
- ↑ Craigie R, Bushman FD (xullo de 2012). "HIV DNA integration". Cold Spring Harbor Perspectives in Medicine 2 (7): a006890. PMC 3385939. PMID 22762018. doi:10.1101/cshperspect.a006890.
- ↑ Burdick RC, Pathak VK (xaneiro de 2021). "Efficient HIV-1 in vitro reverse transcription: optimal capsid stability is required". Signal Transduction and Targeted Therapy 6 (1): 13. PMC 7804106. PMID 33436564. doi:10.1038/s41392-020-00458-3.
- ↑ Maldarelli F (febreiro de 2016). "The role of HIV integration in viral persistence: no more whistling past the proviral graveyard". The Journal of Clinical Investigation 126 (2): 438–447. PMC 4731194. PMID 26829624. doi:10.1172/JCI80564.
- ↑ 11,0 11,1 11,2 Mahboubi-Rabbani M, Abbasi M, Hajimahdi Z, Zarghi A (2021). "HIV-1 Reverse Transcriptase/Integrase Dual Inhibitors: A Review of Recent Advances and Structure-activity Relationship Studies". Iranian Journal of Pharmaceutical Research 20 (2): 333–369. PMC 8457747. PMID 34567166. doi:10.22037/ijpr.2021.115446.15370.
Véxase tamén
[editar | editar a fonte]Outros artigos
[editar | editar a fonte]Bibliografía
[editar | editar a fonte]- Maertens GN, Engelman AN, Cherepanov P (xaneiro de 2022). "Structure and function of retroviral integrase". Nature Reviews. Microbiology 20 (1): 20–34. PMC 8671357. PMID 34244677. doi:10.1038/s41579-021-00586-9.
Ligazóns externas
[editar | editar a fonte]- PDB-101 Molecule of the Month: 135 HIV Integrase
- Integrases Medical Subject Headings (MeSH) na Biblioteca Nacional de Medicina dos EUA.
