Man EF
| Man EF | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Estrutura da calmodulina de Paramecium tetraurelia recombinante.[1] | |||||||||
| Identificadores | |||||||||
| Símbolo | efhand | ||||||||
| Pfam | PF00036 | ||||||||
| InterPro | IPR002048 | ||||||||
| PROSITE | PDOC00018 | ||||||||
| SCOPe | 1osa / SUPFAM | ||||||||
| OPM protein | 1djx | ||||||||
| CDD | cd00051 | ||||||||
| |||||||||
A man EF (en inglés EF hand) é un dominio estrutural ou morivo de tipo hélice-bucle-hélice que se encontra nunha gran familia de proteínas que se unen ao calcio.
O motivo man EF ten unha topoloxía hélice-bucle-hélice, moi similar á que teñen os dedos índice e polgar estendidos da man humana (de aí o seu nome), na cal os ións Ca2+ son coordinados por ligandos situados no bucle. O motivo debe o seu nome á nomenclatura tradicional usada para describir a proteína parvalbumina, un de cuxos dominios se chama EF; esta proteína contén tres motivos man EF e está probablemente implicada na relaxación muscular por medio da súa actividade de unión ao calcio.
A man EF consta de dúas hélices alfa unidas por unha curta rexión bucle (normalmente de 12 aminoácidos), que xeralmente se une ao calcio. As mans EF tamén aparecen, por exemplo, en cada un dos dominios estruturais da proteína de sinalización calmodulina e na proteína muscular troponina-C.
Sitio de unión ao ión calcio
[editar | editar a fonte]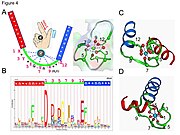

- Na man EF O ión calcio está coordinado nunha configuración bipiramidal pentagonal. Os seis residuos implicados na unión están nas posicións 1, 3, 5, 7, 9 e 12; estes residuos denomínanse X, Y, Z, -Y, -X e -Z. Os residuosde invariables Glu ou Asp na posición 12 proporcionan dous oxíxenos para ligar o calcio (ligando bidentado).
- O ión calcio está unido polos átomos do esqueleto da molécula proteica e polas cadeas laterais de aminoácidos, especificamente as dos residuos de aminoácidos de tipo ácido aspartato e glutamato. Estes residuos están cargados negativamente nas súas cadeas laterais e establecerán unha interacción de carga co ión calcio cargado positivamente. O motivo man EF foi un dos motivos estruturais nos que se analizaron en detalle os seus requirimentos de secuencia. Cinco dos residuos do bucle únense ao calcio e teñen unha forte preferencia polas cadeas laterais que conteñen oxíxeno, especialmente o aspartato e glutamato. O sexto residuo do bucle é necesariamente a glicina debido a esixencias conformacionais do esqueleto da proteína. Os residuos restantes son tipicamente hidrofóbicos e forman un núcleo hidrofóbico que se une e estabiliza as dúas hélices.
- Despois de unirse ao Ca2+, este motivo pode sufrir cambios conformacionais que permiten as funcións reguladas por Ca2+ como se ven nos efectores de Ca2+ como a calmodulina (CaM) e a troponina C (TnC) e nos tampóns de Ca2+ como a calreticulina e a calbindina D9k. Aínda que a maioría das proteínas de unión ao calcio de man EF (CaBPs) conteñen motivos man EF por pares, descubríronse tamén proteínas de unión ao calcio con mans EF sinxelas (non por pares) tanto en bacterias coma en eucariotas. Ademais, uns motivos similares chamados "motivos de tipo man EF" (EF-hand-like motifs) encontráronse en varias bacterias. Aínda que as propiedades de coordinación seguen sendo similares ás do motivo de man EF de hélice-bucle-hélice de 29 residuos canónico, o motivo similar de "tipo man EF" difire das "mans EF" en que contén desviacións na súa estrutura secundaria nas secuencias flanqueantes e/ou unha variación na lonxitude do bucle que coordina ao Ca2+.
Predición
[editar | editar a fonte]
A busca de patróns (sinatura motivo) é unha das maneiras máis directas de predicir sitios de unión ao Ca2+ man EF continuos nas proteínas. Xerouse un patrón PS00018 para predicir mans EF canónicas, basándose nos resultados de aliñamentos de secuencias de motivos man EF canónicos, especialmente nas cadeas laterais conservadas directamente implicadas na unión ao Ca2+. Poden atoparse servidores de predición na sección de ligazóns externas.
Clasificación
[editar | editar a fonte]- Desde a delineación do motivo man EF en 1973, a familia de proteínas de man EF ampliouse para incluír polo menos 66 subfamilias ata agora. Os motivos man EF divídense en dous grandes grupos: as mans EF canónicas e as pseudo-mans EF:
- Mans EF canónicas como as que teñen a calmodulina (CaM) e a proteína procariótica de tipo CaM caleritrina. O bucle de man EF canónico de 12 residuos únese ao Ca2+ principalmente por medio de carboxilatos ou carbonilos das cadeas laterais (posicións na secuencia do bucle 1, 3, 5, 12). O residuo no eixe –X coordina o ión Ca2+ a través dunha molécula de auga que fai de ponte. O bucle man EF ten un ligando bidentado (Glu ou Asp) no eixe –Z.
- Pseudo-mans EF, que se encontran exclusivamente no N-terminal de proteínas do tipo das S100 e S100. Os bucles de pseudo-man EF de 14 residuos quelan o Ca2+ principalmente por vía dos carbonilos do esqueleto da molécula proteica (nas posicións 1, 4, 6, 9).
Cuestións adicionais:

- Informouse en bacterias e virus da existencia de proteínas "de tipo man EF" (EF-hand-like proteins) con elementos estruturais flanqueantes diversificados arredor do bucle de unión ao Ca2+. Estas proteínas de "tipo man EF" procarióticas están moi implicadas na sinalización e homeostase do Ca2+ en bacterias. Conteñen lonxitudes flexibles do bucle de unión ao Ca2+ que difiren dos motivos man EF. Con todo, as súas propiedades de coordinación lembran os motivos man EF clásicos.
- Por exemplo, o sito de unión ao Ca2+ semicontinuo na proteína de unión á D-galactosa (GBP) contén un bucle de nove residuos. O ión Ca2+ está coordinado por sete átomos de oxíxeno da proteína, cinco dos cales son do bucloe que imita a man EF canónica, mentres que os outros dous son do grupo carboxilato dun glutamato distante.
- Outro exemplo é un novo dominio chamado Excalibur (rexión de unión ao Ca2+ extracelular) illado de Bacillus subtilis. Este dominio ten un bucle de unión ao Ca2+ de 10 residuos conservados sorprendentemente similar ao do bucle de man EF de 12 residuos canónico.
- A diversidade da estrutura da rexión flanqueante é ilustrada polo descubrimento de dominios de "tipo man EF" en proteínas bacterianas. Por exemplo, unha hélice-bucle-febra e vez da estrutura hélice-bucle-hélice está presente na proteína de unión á galactosa periplásmica (de Salmonella typhimurium, PDB 1gcg) ou na proteína de unión ao alxinato (Sphingomonas sp., 1kwh); outros exemplos son que a hélice entrante está ausente no antíxeno protector (de Bacillus anthracis, 1acc) ou na dockerina (Clostridium thermocellum, 1daq).
- Entre todas as estruturas coñecidas ata agora, a maioría dos motivos man EF aparecen por pares formados por dúas EF canónicas ou por un motivo pseudo-man e outro canónico. En proteínas con números impares de mans EF, como a calpaína penta-man EF, os motivos man EF estaban acoplados por medio de homo ou heterodimerización. As proteínas identificadas recentemente proteína sensora do Ca2+ ER que contén a man EF, e as moléculas de interacción estromal 1 e 2 (STIM1, STIM2), conteñen un motivo man EF canónico de unión ao Ca2+ que se aparea con outro inmediato, que é unha man EF de unión non-ao-Ca2+ "agochada" atipicamente augas abaixo. Os motivos man EF sinxelos (nn pareados) poden servir como módulos para o atraque de proteínas: por exemplo, a man EF sinxela das proteínas NKD1 e NKD2 únese ás proteínas Dishevelled (DVL1, DVL2, DVL3).
- Funcionalmente, as mans EF poden dividirse en dúas clases: 1) proteínas de sinalización, e 2) proteínas de tamponamento/transporte. O primeiro grupo é o máis grande e inclúe os membros mellor coñecidos da familia como a calmodulina, troponina C e S100B. Estas proteínas sofren tipicamente un cambio conformacional dependente do calcio que abre un sitio de unión á diana. O último grupo é o representado pola calbindina D9k, que non sofre cambios conformacionais dependentes do calcio.
Subfamilias
[editar | editar a fonte]Exemplos
[editar | editar a fonte]Aequorina
[editar | editar a fonte]A aequorina é unha proteína de unión ao calcio illada do cnidario Aequorea victoria (de onde lle vén o nome). A aequorina pertence á familia de man EF de proteínas de unión ao calcio (CaBPs), con bucles de man EF que están moi relacionados coas proteínas de unión ao calcio de mamíferos. Ademais, a aequorina foi utilizada durante anos como indicador de Ca2+ e demostrouse que é segura e ben tolerada polas células. A aequorina está constituçida por dous compoñentes: o compoñente de unión ao calcio apoaequorina (AQ) e a molécula quimioluminescente coelenterazina. A porción AQ desta porteína contén os dominios de unión ao calcio man EF.[2]
Proteínas humanas
[editar | editar a fonte]Proteínas humanas que conteñan este motivo son as seguintes:
- ACTN1; ACTN2; ACTN3; ACTN4; APBA2BP; AYTL1; AYTL2
- C14orf143; CABP1; CABP2; CABP3; CABP4; CABP5; CABP7; CALB1; CALB2; CALM2; CALM3; CALML3; CALML4; CALML5; CALML6; CALN1; CALU; CAPN1; CAPN11; CAPN2; CAPN3; CAPN9; CAPNS1; CAPNS2; CAPS; CAPS2; CAPSL; CBARA1; CETN1; CETN2; CETN3; CHP; CHP2; CIB1; CIB2; CIB3; CIB4; CRNN
- DGKA; DGKB; DGKG; DST; DUOX1; DUOX2
- EFCAB1; EFCAB2; EFCAB4A; EFCAB4B; EFCAB6; EFCBP1; EFCBP2; EFHA1; EFHA2; EFHB; EFHC1; EFHD1; EFHD2; EPS15; EPS15L1
- FKBP10; FKBP14; FKBP7; FKBP9; FKBP9L; FREQ; FSTL1; FSTL5
- GCA; GPD2; GUCA1A; GUCA1B; GUCA1C
- hippocalcina; HPCAL1; HPCAL4; HZGJ
- IFPS; ITSN1; ITSN2; KCNIP1; KCNIP2; KCNIP3; KCNIP4; KIAA1799
- LCP1
- MACF1; MRLC2; MRLC3; MST133; MYL1; MYL2; MYL5; MYL6B; MYL7; MYL9; MYLC2PL; MYLPF
- NCALD; NIN; NKD1; NKD2; NLP; NOX5; NUCB1; NUCB2
- OCM
- PDCD6; PEF1; PKD2; PLCD1; PLCD4; PLCH1; PLCH2; PLS1; PLS3; PP1187; PPEF1; PPEF2; PPP3R1; PPP3R2; PRKCSH; PVALB
- RAB11FIP3; RASEF; RASGRP; RASGRP1; RASGRP2; RASGRP3; RCN1; RCN2; RCN3; RCV1; RCVRN; REPS1; RHBDL3; RHOT1; RHOT2; RPTN; RYR2; RYR3
- S100A1; S100A11; S100A12; S100A6; S100A8; S100A9; S100B; S100G; S100Z; SCAMC-2; SCGN; SCN5A; SDF4; SLC25A12; SLC25A13; SLC25A23; SLC25A24; SLC25A25; SPATA21; SPTA1; SPTAN1; SRI
- TBC1D9; TBC1D9B; TCHH; TESC; TNNC1; TNNC2
- USP32
- VSNL1
- ZZEF1
Notas
[editar | editar a fonte]- ↑ Ban C, Ramakrishnan B, Ling KY, Kung C, Sundaralingam M (xaneiro de 1994). "Structure of the recombinant Paramecium tetraurelia calmodulin at 1.68 A resolution". Acta Crystallogr. D Biol. Crystallogr. 50 (Pt 1): 50–63. PMID 15299476. doi:10.1107/S0907444993007991.
- ↑ Detert JA, Adams EL, Lescher JD, Lyons JA, Moyer JR (2013). "Pretreatment with Apoaequorin Protects Hippocampal CA1 Neurons from Oxygen-Glucose Deprivation". PLoS ONE 8 (11): e79002. PMC 3823939. PMID 24244400. doi:10.1371/journal.pone.0079002.
Véxase tamén
[editar | editar a fonte]Outros artigos
[editar | editar a fonte]- Dockerina (outro motivo de unión ao calcio composto por hélices alfa)
Bibliografía
[editar | editar a fonte]- Branden C, Tooze J (1999). "Chapter 2: Motifs of protein structure". Introduction to Protein Structure. New York: Garland Pub. pp. 24–25. ISBN 0-8153-2305-0.
- Nakayama S, Kretsinger RH (1994). "Evolution of the EF-hand family of proteins". Annu Rev Biophys Biomol Struct 23: 473–507. PMID 7919790. doi:10.1146/annurev.bb.23.060194.002353.
- Zhou Y, Yang W, Kirberger M, Lee HW, Ayalasomayajula G, Yang JJ (novembro de 2006). "Prediction of EF-hand calcium-binding proteins and analysis of bacterial EF-hand proteins". Proteins 65 (3): 643–55. PMID 16981205. doi:10.1002/prot.21139.
- Zhou Y, Frey TK, Yang JJ (xullo de 2009). "Viral calciomics: interplays between Ca2+ and virus". Cell Calcium 46 (1): 1–17. PMC 3449087. PMID 19535138. doi:10.1016/j.ceca.2009.05.005.
- Nakayama S, Moncrief ND, Kretsinger RH (maio de 1992). "Evolution of EF-hand calcium-modulated proteins. II. Domains of several subfamilies have diverse evolutionary histories". J. Mol. Evol. 34 (5): 416–48. PMID 1602495. doi:10.1007/BF00162998.
- Hogue CW, MacManus JP, Banville D, Szabo AG (xullo de 1992). "Comparison of terbium (III) luminescence enhancement in mutants of EF hand calcium binding proteins". J. Biol. Chem. 267 (19): 13340–7. PMID 1618836.
- Bairoch A, Cox JA (setembro de 1990). "EF-hand motifs in inositol phospholipid-specific phospholipase C". FEBS Lett. 269 (2): 454–6. PMID 2401372. doi:10.1016/0014-5793(90)81214-9.
- Finn BE, Forsén S (xaneiro de 1995). "The evolving model of calmodulin structure, function and activation". Structure 3 (1): 7–11. PMID 7743133. doi:10.1016/S0969-2126(01)00130-7.
- Stathopulos PB, Zheng L, Li GY, Plevin MJ, Ikura M (outubro de 2008). "Structural and mechanistic insights into STIM1-mediated initiation of store-operated calcium entry". Cell 135 (1): 110–22. PMID 18854159. doi:10.1016/j.cell.2008.08.006.
- Nelson MR, Thulin E, Fagan PA, Forsén S, Chazin WJ (febreiro de 2002). "The EF-hand domain: a globally cooperative structural unit". Protein Sci. 11 (2): 198–205. PMC 2373453. PMID 11790829. doi:10.1110/ps.33302.
Ligazóns externas
[editar | editar a fonte]- Eukaryotic Linear Motif resource: motivo de tipo LIG_EH_1
- Eukaryotic Linear Motif resource: motivo de tipo LIG_IQ
- Eukaryotic Linear Motif resource: motivo de tipo DOC_PP2B_LxvP_1
- Eukaryotic Linear Motif resource: motivo de tipo LIG_IQ
- Nelson M, Chazin W. "EF-Hand Calcium-Binding Proteins Data Library". Vanderbilt University. Arquivado dende o orixinal o 25 de agosto de 2009. Consultado o 2009-08-29.
- Haiech J. "EF-hand protein database (EF-handome)". European Calcium Society and the Université Libre de Bruxelles. Arquivado dende o orixinal o 09 de agosto de 2009. Consultado o 2009-08-29.
upon request to haiech@pharma.u-strasbg.fr
- Yang J. "Calciomics". Georgia State University. Consultado o 2009-08-29.
prediction server for EF-hand calcium binding proteins
