MGMT (proteína)
MGMT
| |
| Identificadores | |
| Símbolo | MGMT |
| Símbolos alt. | Mgmt, AGT, AI267024, Agat |
| Entrez | 4255 |
| RefSeq | XP_005252739.1 |
| UniProt | P16455 |
| Outros datos | |
| Locus | Cr. 10 :(129.47 – 129.77 Mb) |
A O6-metilguanina ADN metiltransferase (MGMT), tamén chamada O6-alquilguanina ADN alquiltransferase (AGT ou AGAT) é unha proteína que nos humanos está codificada no xene MGMT do cromosoma 10.[1][2] A O6-metilguanina ADN metiltransferase é esencial para a estabilidade do xenoma. Repara as lesións no ADN mutaxénicas que ocorren de forma natural que orixinan O6-metilguanina (tamén chamada 6-O-metilguanina), converténdoa de novo en guanina e impide a discordancia e os erros durante a replicación do ADN e a transcrición. En consecuencia, a perda de MGMT incrementa o risco de cancro en ratos despois da exposición a axentes alquilantes.[3] Os dous isocimas bacterianos equivalentes son Ada e Ogt.
Función e mecanismo
[editar | editar a fonte]| O6-alquilguanina ADN alquiltransferase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| Número EC | 2.1.1.63 | ||||||||
| Número CAS | 77271-19-3 | ||||||||
| Bases de datos | |||||||||
| IntEnz | vista de IntEnz | ||||||||
| BRENDA | entrada de BRENDA | ||||||||
| ExPASy | vista de NiceZyme | ||||||||
| KEGG | entrada de KEGG | ||||||||
| MetaCyc | vía metabólica | ||||||||
| PRIAM | perfil | ||||||||
| Estruturas PDB | RCSB PDB PDBe PDBj PDBsum | ||||||||
| |||||||||
Aínda que os mutáxenos alquilantes modifican preferencialmente a base guanina na posición N7, a formación de O6-alquil-guanina é unha lesión carcinoxénica importante no ADN. Este aduto do ADN é retirado pola proteína de reparación O6-alquilguanina ADN alquiltransferase por medio dun mecanismo SN2. Esta proteína non é un verdadeiro encima, xa que elimina o grupo alquilo da lesión nunha reacción estoquiométrica e o encima activo non se rexenera despois de que é alquilado (o que se denomina ás veces suicidio dun encima). O residuo aceptor de metilos na proteína é unha cisteína.[4]
- Desmetilación da 6-O-metilguanosina a guanosina
Importancia clínica
[editar | editar a fonte]A metilación dos promotores dos xenes pode xogar un papel significativo na carcinoxénese. En pacientes con glioblastoma multiforme, un tipo grave de tumor cerebral, o estado de metilación do xene MGMT determina se as células tumorais responderán ao tratamento con temozolomida; se o promotor está metilado, a temozolomida é máis efectiva.[5] A nivel clínico, isto trradúcese nunha supervivencia prolongada de pacientes de glioblastoma cun promotor MGMT metilado. Ademais, a metilación de MGMT pode utilizarse para predicir a supervivencia do paciente en modelos de predición clínica.[6] Para comprobar o estado de metilación do promotor de MGMT en instalacións clínicas, prefírense os métodos baseados no ADN, como a reacción en cadea da polimerase específica de metilación (MS-PCR) ou a pirosecuenciación, mellor que ensaios inmunohistoquímicos ou baseados no ARN.[7]
O xene MGMT é tamén unha útil ferramenta para incrementar a eficacia da terapia xénica. Ao usar un segundo vector de dous compoñentes consistente nun transxene de interese e o MGMT, pode utilizarse a selección de fármacos in vivo para seleccionar células que foron transducidas con éxito.[8]
Os mutáxenos presentes no ambiente,[9] no fume do tabaco[10] e alimentos,[11] así como os produtos metabólicos endóxenos[12] xeran especies electrófilas reactivas que alquilan ou metilan o ADN especificamente, xerando 6-O-metilguanina (m6G).
En 1985 Yarosh resumiu os traballos iniciais que estableceron a m6G como a base alquilada no ADN que era a máis mutaxénica e carcinoxénica.[13] En 1994 Rasouli-Nia et al.[14] mostraron que aproximadamente se inducía unha mutación por cada oito m6Gs que non eran reparadas no ADN. As mutacións poden causar a progresión cara ao cancro por un proceso de selección natural.[15]
Expresión no cancro
[editar | editar a fonte]| Tipo de cancro | Frecuencia da deficiencia no cancro | Frecuencia da deficiencia en defectos de campo adxacentes |
|---|---|---|
| Cervical[16] | 61% | 39% |
| Colorrectal | 40%-90%[17][18][19][20][21] | 11%-34%[17][18] |
| Colorrectal con inestabilidade de microsatélites[22] | 70% | 60% |
| Adenocarcinoma esofáxico | 71%-79%[23][24] | 89%[24] |
| Carcinoma de células escamosas esofáxico | 38%-96%[23][25][26][26] | 65%[26] |
| Glioblastoma debido á metilación do promotor | 44%-59%[27][28] | |
| Carcinoma de células escamosas de cabeza e pescozo | 54%[29] | |
| Carcinoma hepatocelular (asociado ao virus da hepatite C)[30] | 68% | 65% |
| Larinxe | 54%-61%[31][32] | 38%[32] |
| Estómago | 32%-88%[33][34][34] | 17%-78%[33][34][34] |
| Tiroide[35] | 87% |
Represión epixenética
[editar | editar a fonte]Só unha minoría dos cancros esporádicos cunha deficiencia na reparación do ADN teñen unha mutación nun xene de reparación do ADN. Porén, unha maioría dos cancros esporádicos con deficiencia na reparación do ADN si teñen unha ou máis alteracións epixenéticas, que reducen ou silencian a expresión de xenes de reparación do ADN. Por exemplo, nun estudo de 113 casos de cancro colorrectal secuencial, só catro tiñan unha mutación con cambio de sentido no xene de reparación do ADN MGMT, mentres que a maioría tiñan reducida a expresión de MGMT debido á metilación da rexión promotora de MGMT (o que é unha alteración epixenética).[36]
MGMT pode ser reprimido epixeneticamente de diversas maneiras.[37] Cando a expresión de MGMT está reprimida en cancros, isto xeralmente débese á metilación da súa rexión promotora.[37] Porén, a expresión pode tamén ser reprimida por dimetilación da lisina 9 da histona H3[38] ou por sobreexpresión de diversos microARNs como miR-181d, miR-767-3p e miR-603.[37][39]
Deficiencia en defectos de campo
[editar | editar a fonte]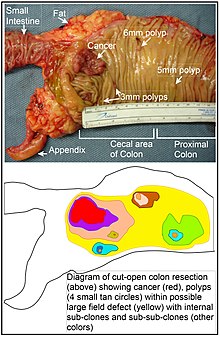
Un defecto de campo é unha área ou "campo" de epitelio que foi precondicionado debido a cambios epixenéticos ou mutacións de modo que predispón cara ao desenvolvemento dun cancro. Un defecto de campo ilústrase na foto e diagrama mostrado dun segmento de colon con cancro de colon e catro pequenos pólipos na mesma área. Como indicou Rubin, "A gran maioría dos estudos en investigación do cancro fixéronse en tumores ben definidos in vivo ou en focos neoplásicos discretos in vitro.[40] Porén, hai probas de que máis do 80% das mutacións somáticas que se encontran en tumores colorrectais humanos de fenotipo mutador ocorren antes do comezo da expansión clonal terminal."[41] De xeito similar, Vogelstein et al.[42] sinalaron que máis da metde das mutacións somáticas identificadas en tumores ocorren na fase preneoplásica (nun defecto de campo), durante o crecemento de células aparentemente normais.
Na táboa de arriba indícase que se detectaron deficiencias de MGMT nos defectos de campo (en tecidos histoloxicamente normais) que rodean a maior parte dos cancros. Se a MGMT se reduce ou silencia epixeneticamente, probablemente non conferirá unha vantaxe selectiva a unha célula nai. Porén, a expresión reducida ou ausente de MGMT causará un incremento das taxas de mutación, e un ou máis dos xenes mutados poden darlle á célula unha vantaxe selectiva. O xene MGMT con expresión deficiente podería despois ser portado polas células como un xene pasaxeiro (autostopista) selectivamente neutro ou só lixeiramente deletéreo cando a célula nai mutada xere un clon expandido. A presenza continuada dun clon cun xene MGMT reprimido epixeneticamente continuaría xerando máis mutacións, algunhas das cales producirían o tumor.
Deficiencia con danos exóxenos
[editar | editar a fonte]A deficiencia de MGMT por si soa pode non ser suficiente para causar a progesión do cancro. Os ratos con mutación homocigota en MGMT non desenvolven máis cancros que os ratos de tipo salvaxe cando crecen sen estrés.[43] Porén, o tratamento estresante dos ratos con azoximetano e dextrán sulfato causaba máis de catro tumores de colon por rato mutante para MGMT, pero menos dun tumor por rato de tipo salvaxe.[44]
Represión en coordinación con outros xenes de reparación do ADN
[editar | editar a fonte]Nos cancros, con frecuencia moitos xenes de reparación do ADN están reprimidos simultaneamente.[45] Nun exemplo, que implica ao xene MGMT, Jiang et al.[46] realizaron un estudo no que avaliaron a expresión dos ARNm de 27 xenes de reparación do ADN en 40 astrocitomas comparados con tecidos cerebrais normais de individuos sen astrocitoma. Entre os 27 xenes de reparación do ADN avaliados, 13 deles, que eran: MGMT, NTHL1, OGG1, SMUG1, ERCC1, ERCC2, ERCC3, ERCC4, MLH1, MLH3, RAD50, XRCC4 e XRCC5, estaban significativamene regulados á baixa en cada un dos tres graos (II, III e IV) dos astrocitomas. A represión destes 13 xenes tanto en astrocitomas de baixo grao coma de maior grao suxeriu que poden ser importantes nos estadios temperán e posteriores dos astrocitomas. Noutro exemplo, Kitajima et al.[47] atoparon que a inmunorreactividade para a expresión de MGMT e MLH1 estaba estreitamente correlacionada en 135 espécimes de cancro gástrico e a perda de MGMT e hMLH1 parecía estar acelarada sincronicamente durante a progresión de tumores.
A expresión deficiente de múltiples xenes de reparación do ADN obsérvase con frecuencia nos cancros,[45] e pode contribuír a que se produzan miles de mutacións que xeralmente se encontran nos cancros.
Interaccións
[editar | editar a fonte]A O6-metilguanina ADN metiltransferase presenta interaccións co receptor de estróxenos alfa.[48]
Notas
[editar | editar a fonte]- ↑ Tano K, Shiota S, Collier J, Foote RS, Mitra S (January 1990). "Isolation and structural characterization of a cDNA clone encoding the human DNA repair protein for O6-alkylguanine". Proc. Natl. Acad. Sci. U.S.A. 87 (2): 686–90. PMC 53330. PMID 2405387. doi:10.1073/pnas.87.2.686.
- ↑ Natarajan AT, Vermeulen S, Darroudi F, Valentine MB, Brent TP, Mitra S, Tano K (January 1992). "Chromosomal localization of human O6-methylguanine-DNA methyltransferase (MGMT) gene by in situ hybridization". Mutagenesis 7 (1): 83–5. PMID 1635460. doi:10.1093/mutage/7.1.83.
- ↑ Shiraishi A, Sakumi K, Sekiguchi M (October 2000). "Increased susceptibility to chemotherapeutic alkylating agents of mice deficient in DNA repair methyltransferase". Carcinogenesis 21 (10): 1879–83. PMID 11023546. doi:10.1093/carcin/21.10.1879.
- ↑ Kaina B, Christmann M, Naumann S, Roos WP (August 2007). "MGMT: key node in the battle against genotoxicity, carcinogenicity and apoptosis induced by alkylating agents". DNA Repair (Amst.) 6 (8): 1079–99. PMID 17485253. doi:10.1016/j.dnarep.2007.03.008.
- ↑ Hegi ME, Diserens AC, Gorlia T, Hamou MF, de Tribolet N, Weller M, Kros JM, Hainfellner JA, Mason W, Mariani L, Bromberg JE, Hau P, Mirimanoff RO, Cairncross JG, Janzer RC, Stupp R (2005). "MGMT gene silencing and benefit from temozolomide in glioblastoma". N. Engl. J. Med. 352 (10): 997–1003. PMID 15758010. doi:10.1056/NEJMoa043331.
- ↑ Molenaar RJ, Verbaan D, Lamba S, Zanon C, Jeuken JW, Boots-Sprenger SH, Wesseling P, Hulsebos TJ, Troost D, van Tilborg AA, Leenstra S, Vandertop WP, Bardelli A, van Noorden CJ, Bleeker FE (2014). "The combination of IDH1 mutations and MGMT methylation status predicts survival in glioblastoma better than either IDH1 or MGMT alone". Neuro-oncology 16 (9): 1263–73. PMC 4136888. PMID 24510240. doi:10.1093/neuonc/nou005.
- ↑ Preusser, M.; Janzer, Charles R.; Felsberg, J.; Reifenberger, G.; Hamou, M. F.; Diserens, A. C.; Stupp, R.; Gorlia, T.; Marosi, C.; Heinzl, H.; Hainfellner, J. A.; Hegi, M. (Oct 2008). "Anti-O6-methylguanine-methyltransferase (MGMT) immunohistochemistry in glioblastoma multiforme: observer variability and lack of association with patient survival impede its use as clinical biomarker". Brain Pathol 18 (4): 520–532. doi:10.1111/j.1750-3639.2008.00153.x.
- ↑ Chang AH, Stephan MT, Lisowski L, Sadelain M (2008). "Erythroid-specific human factor IX delivery from in vivo selected hematopoietic stem cells following nonmyeloablative conditioning in hemophilia B mice". Mol. Ther. 16 (10): 1745–52. PMC 2658893. PMID 18682698. doi:10.1038/mt.2008.161.
- ↑ Bartsch H, Montesano R (1984). "Relevance of nitrosamines to human cancer". Carcinogenesis 5 (11): 1381–93. PMID 6386215. doi:10.1093/carcin/5.11.1381.
- ↑ Christmann M, Kaina B (2012). "O(6)-methylguanine-DNA methyltransferase (MGMT): impact on cancer risk in response to tobacco smoke". Mutat. Res. 736 (1-2): 64–74. PMID 21708177. doi:10.1016/j.mrfmmm.2011.06.004.
- ↑ Fahrer J, Kaina B (2013). "O6-methylguanine-DNA methyltransferase in the defense against N-nitroso compounds and colorectal cancer". Carcinogenesis 34 (11): 2435–42. PMID 23929436. doi:10.1093/carcin/bgt275.
- ↑ De Bont R, van Larebeke N (2004). "Endogenous DNA damage in humans: a review of quantitative data". Mutagenesis 19 (3): 169–85. PMID 15123782. doi:10.1093/mutage/geh025.
- ↑ Yarosh DB (1985). "The role of O6-methylguanine-DNA methyltransferase in cell survival, mutagenesis and carcinogenesis". Mutat. Res. 145 (1-2): 1–16. PMID 3883145. doi:10.1016/0167-8817(85)90034-3.
- ↑ Rasouli-Nia A, Sibghat-Ullah, Mirzayans R, Paterson MC, Day RS (1994). "On the quantitative relationship between O6-methylguanine residues in genomic DNA and production of sister-chromatid exchanges, mutations and lethal events in a Mer- human tumor cell line". Mutat. Res. 314 (2): 99–113. PMID 7510369. doi:10.1016/0921-8777(94)90074-4.
- ↑ Bernstein C, Prasad AR, Nfonsam V, Bernstein H. (2013). DNA Damage, DNA Repair and Cancer, New Research Directions in DNA Repair, Prof. Clark Chen (Ed.), ISBN 978-953-51-1114-6, InTech, http://www.intechopen.com/books/new-research-directions-in-dna-repair/dna-damage-dna-repair-and-cancer
- ↑ Iliopoulos D, Oikonomou P, Messinis I, Tsezou A (2009). "Correlation of promoter hypermethylation in hTERT, DAPK and MGMT genes with cervical oncogenesis progression". Oncol. Rep. 22 (1): 199–204. PMID 19513524. doi:10.3892/or_00000425.
- ↑ 17,0 17,1 Shen L, Kondo Y, Rosner GL, Xiao L, Hernandez NS, Vilaythong J, Houlihan PS, Krouse RS, Prasad AR, Einspahr JG, Buckmeier J, Alberts DS, Hamilton SR, Issa JP (2005). "MGMT promoter methylation and field defect in sporadic colorectal cancer". J. Natl. Cancer Inst. 97 (18): 1330–8. PMID 16174854. doi:10.1093/jnci/dji275.
- ↑ 18,0 18,1 Lee KH, Lee JS, Nam JH, Choi C, Lee MC, Park CS, Juhng SW, Lee JH (2011). "Promoter methylation status of hMLH1, hMSH2, and MGMT genes in colorectal cancer associated with adenoma-carcinoma sequence". Langenbecks Arch Surg 396 (7): 1017–26. PMID 21706233. doi:10.1007/s00423-011-0812-9.
- ↑ Psofaki V, Kalogera C, Tzambouras N, Stephanou D, Tsianos E, Seferiadis K, Kolios G (2010). "Promoter methylation status of hMLH1, MGMT, and CDKN2A/p16 in colorectal adenomas". World J. Gastroenterol. 16 (28): 3553–60. PMC 2909555. PMID 20653064. doi:10.3748/wjg.v16.i28.3553.
- ↑ Amatu A, Sartore-Bianchi A, Moutinho C, Belotti A, Bencardino K, Chirico G, Cassingena A, Rusconi F, Esposito A, Nichelatti M, Esteller M, Siena S (2013). "Promoter CpG island hypermethylation of the DNA repair enzyme MGMT predicts clinical response to dacarbazine in a phase II study for metastatic colorectal cancer". Clin. Cancer Res. 19 (8): 2265–72. PMID 23422094. doi:10.1158/1078-0432.CCR-12-3518.
- ↑ Mokarram P, Zamani M, Kavousipour S, Naghibalhossaini F, Irajie C, Moradi Sarabi M, Hosseini SV (2013). "Different patterns of DNA methylation of the two distinct O6-methylguanine-DNA methyltransferase (O6-MGMT) promoter regions in colorectal cancer". Mol. Biol. Rep. 40 (5): 3851–7. PMID 23271133. doi:10.1007/s11033-012-2465-3.
- ↑ Svrcek M, Buhard O, Colas C, Coulet F, Dumont S, Massaoudi I, Lamri A, Hamelin R, Cosnes J, Oliveira C, Seruca R, Gaub MP, Legrain M, Collura A, Lascols O, Tiret E, Fléjou JF, Duval A (2010). "Methylation tolerance due to an O6-methylguanine DNA methyltransferase (MGMT) field defect in the colonic mucosa: an initiating step in the development of mismatch repair-deficient colorectal cancers". Gut 59 (11): 1516–26. PMID 20947886. doi:10.1136/gut.2009.194787.
- ↑ 23,0 23,1 Hasina R, Surati M, Kawada I, Arif Q, Carey GB, Kanteti R, Husain AN, Ferguson MK, Vokes EE, Villaflor VM, Salgia R (2013). "O-6-methylguanine-deoxyribonucleic acid methyltransferase methylation enhances response to temozolomide treatment in esophageal cancer". J Carcinog 12: 20. PMC 3853796. PMID 24319345. doi:10.4103/1477-3163.120632.
- ↑ 24,0 24,1 Kuester D, El-Rifai W, Peng D, Ruemmele P, Kroeckel I, Peters B, Moskaluk CA, Stolte M, Mönkemüller K, Meyer F, Schulz HU, Hartmann A, Roessner A, Schneider-Stock R (2009). "Silencing of MGMT expression by promoter hypermethylation in the metaplasia-dysplasia-carcinoma sequence of Barrett's esophagus". Cancer Lett. 275 (1): 117–26. PMC 4028828. PMID 19027227. doi:10.1016/j.canlet.2008.10.009.
- ↑ Ling ZQ, Li P, Ge MH, Hu FJ, Fang XH, Dong ZM, Mao WM (2011). "Aberrant methylation of different DNA repair genes demonstrates distinct prognostic value for esophageal cancer". Dig. Dis. Sci. 56 (10): 2992–3004. PMID 21674174. doi:10.1007/s10620-011-1774-z.
- ↑ 26,0 26,1 26,2 Su Y, Yin L, Liu R, Sheng J, Yang M, Wang Y, Pan E, Guo W, Pu Y, Zhang J, Liang G (2014). "Promoter methylation status of MGMT, hMSH2, and hMLH1 and its relationship to corresponding protein expression and TP53 mutations in human esophageal squamous cell carcinoma". Med. Oncol. 31 (2): 784. PMID 24366688. doi:10.1007/s12032-013-0784-4.
- ↑ Morandi L, Franceschi E, de Biase D, Marucci G, Tosoni A, Ermani M, Pession A, Tallini G, Brandes A (2010). "Promoter methylation analysis of O6-methylguanine-DNA methyltransferase in glioblastoma: detection by locked nucleic acid based quantitative PCR using an imprinted gene (SNURF) as a reference". BMC Cancer 10: 48. PMC 2843669. PMID 20167086. doi:10.1186/1471-2407-10-48.
- ↑ Quillien V, Lavenu A, Karayan-Tapon L, Carpentier C, Labussière M, Lesimple T, Chinot O, Wager M, Honnorat J, Saikali S, Fina F, Sanson M, Figarella-Branger D (2012). "Comparative assessment of 5 methods (methylation-specific polymerase chain reaction, MethyLight, pyrosequencing, methylation-sensitive high-resolution melting, and immunohistochemistry) to analyze O6-methylguanine-DNA-methyltranferase in a series of 100 glioblastoma patients". Cancer 118 (17): 4201–11. PMID 22294349. doi:10.1002/cncr.27392.
- ↑ Koutsimpelas D, Pongsapich W, Heinrich U, Mann S, Mann WJ, Brieger J (2012). "Promoter methylation of MGMT, MLH1 and RASSF1A tumor suppressor genes in head and neck squamous cell carcinoma: pharmacological genome demethylation reduces proliferation of head and neck squamous carcinoma cells". Oncol. Rep. 27 (4): 1135–41. PMC 3583513. PMID 22246327. doi:10.3892/or.2012.1624.
- ↑ Zekri AR, Bahnasy AA, Shoeab FE, Mohamed WS, El-Dahshan DH, Ali FT, Sabry GM, Dasgupta N, Daoud SS (2014). "Methylation of multiple genes in hepatitis C virus associated hepatocellular carcinoma". J Adv Res 5 (1): 27–40. PMC 4294722. PMID 25685469. doi:10.1016/j.jare.2012.11.002.
- ↑ Pierini S, Jordanov SH, Mitkova AV, Chalakov IJ, Melnicharov MB, Kunev KV, Mitev VI, Kaneva RP, Goranova TE (2014). "Promoter hypermethylation of CDKN2A, MGMT, MLH1, and DAPK genes in laryngeal squamous cell carcinoma and their associations with clinical profiles of the patients". Head Neck 36 (8): 1103–8. PMID 23804521. doi:10.1002/hed.23413.
- ↑ 32,0 32,1 Paluszczak J, Misiak P, Wierzbicka M, Woźniak A, Baer-Dubowska W (2011). "Frequent hypermethylation of DAPK, RARbeta, MGMT, RASSF1A and FHIT in laryngeal squamous cell carcinomas and adjacent normal mucosa". Oral Oncol. 47 (2): 104–7. PMID 21147548. doi:10.1016/j.oraloncology.2010.11.006.
- ↑ 33,0 33,1 Jin J, Xie L, Xie CH, Zhou YF (2014). "Aberrant DNA methylation of MGMT and hMLH1 genes in prediction of gastric cancer". Genet. Mol. Res. 13 (2): 4140–5. PMID 24938706. doi:10.4238/2014.May.30.9.
- ↑ 34,0 34,1 34,2 34,3 Zou XP, Zhang B, Zhang XQ, Chen M, Cao J, Liu WJ (2009). "Promoter hypermethylation of multiple genes in early gastric adenocarcinoma and precancerous lesions". Hum. Pathol. 40 (11): 1534–42. PMID 19695681. doi:10.1016/j.humpath.2009.01.029.
- ↑ Mokhtar M, Kondo K, Namura T, Ali AH, Fujita Y, Takai C, Takizawa H, Nakagawa Y, Toba H, Kajiura K, Yoshida M, Kawakami G, Sakiyama S, Tangoku A (2014). "Methylation and expression profiles of MGMT gene in thymic epithelial tumors". Lung Cancer 83 (2): 279–87. PMID 24388682. doi:10.1016/j.lungcan.2013.12.004.
- ↑ Halford S, Rowan A, Sawyer E, Talbot I, Tomlinson I (June 2005). "O(6)-methylguanine methyltransferase in colorectal cancers: detection of mutations, loss of expression, and weak association with G:C>A:T transitions". Gut 54 (6): 797–802. PMC 1774551. PMID 15888787. doi:10.1136/gut.2004.059535.
- ↑ 37,0 37,1 37,2 Cabrini G, Fabbri E, Lo Nigro C, Dechecchi MC, Gambari R (2015). "Regulation of expression of O6-methylguanine-DNA methyltransferase and the treatment of glioblastoma (Review)". Int. J. Oncol. 47 (2): 417–28. PMID 26035292. doi:10.3892/ijo.2015.3026.
- ↑ Nakagawachi T, Soejima H, Urano T, Zhao W, Higashimoto K, Satoh Y, Matsukura S, Kudo S, Kitajima Y, Harada H, Furukawa K, Matsuzaki H, Emi M, Nakabeppu Y, Miyazaki K, Sekiguchi M, Mukai T (2003). "Silencing effect of CpG island hypermethylation and histone modifications on O6-methylguanine-DNA methyltransferase (MGMT) gene expression in human cancer". Oncogene 22 (55): 8835–44. PMID 14647440. doi:10.1038/sj.onc.1207183.
- ↑ Kushwaha D, Ramakrishnan V, Ng K, Steed T, Nguyen T, Futalan D, Akers JC, Sarkaria J, Jiang T, Chowdhury D, Carter BS, Chen CC (2014). "A genome-wide miRNA screen revealed miR-603 as a MGMT-regulating miRNA in glioblastomas". Oncotarget 5 (12): 4026–39. PMC 4147303. PMID 24994119. doi:10.18632/oncotarget.1974.
- ↑ Rubin H (March 2011). "Fields and field cancerization: the preneoplastic origins of cancer: asymptomatic hyperplastic fields are precursors of neoplasia, and their progression to tumors can be tracked by saturation density in culture". BioEssays 33 (3): 224–31. PMID 21254148. doi:10.1002/bies.201000067.
- ↑ Tsao JL, Yatabe Y, Salovaara R, Järvinen HJ, Mecklin JP, Aaltonen LA, Tavaré S, Shibata D (February 2000). "Genetic reconstruction of individual colorectal tumor histories". Proc. Natl. Acad. Sci. U.S.A. 97 (3): 1236–41. PMC 15581. PMID 10655514. doi:10.1073/pnas.97.3.1236.
- ↑ Vogelstein B, Papadopoulos N, Velculescu VE, Zhou S, Diaz LA, Kinzler KW (March 2013). "Cancer genome landscapes". Science 339 (6127): 1546–58. PMC 3749880. PMID 23539594. doi:10.1126/science.1235122.
- ↑ Meira LB, Calvo JA, Shah D, Klapacz J, Moroski-Erkul CA, Bronson RT, Samson LD (2014). "Repair of endogenous DNA base lesions modulate lifespan in mice". DNA Repair (Amst.) 21: 78–86. PMC 4125484. PMID 24994062. doi:10.1016/j.dnarep.2014.05.012.
- ↑ Wirtz S, Nagel G, Eshkind L, Neurath MF, Samson LD, Kaina B (2010). "Both base excision repair and O6-methylguanine-DNA methyltransferase protect against methylation-induced colon carcinogenesis". Carcinogenesis 31 (12): 2111–7. PMC 2994278. PMID 20732909. doi:10.1093/carcin/bgq174.
- ↑ 45,0 45,1 Bernstein C, Bernstein H (2015). "Epigenetic reduction of DNA repair in progression to gastrointestinal cancer". World J Gastrointest Oncol 7 (5): 30–46. PMC 4434036. PMID 25987950. doi:10.4251/wjgo.v7.i5.30.
- ↑ Jiang Z, Hu J, Li X, Jiang Y, Zhou W, Lu D (2006). "Expression analyses of 27 DNA repair genes in astrocytoma by TaqMan low-density array". Neurosci. Lett. 409 (2): 112–7. PMID 17034947. doi:10.1016/j.neulet.2006.09.038.
- ↑ Kitajima Y, Miyazaki K, Matsukura S, Tanaka M, Sekiguchi M (2003). "Loss of expression of DNA repair enzymes MGMT, hMLH1, and hMSH2 during tumor progression in gastric cancer". Gastric Cancer 6 (2): 86–95. PMID 12861399. doi:10.1007/s10120-003-0213-z.
- ↑ Teo AK, Oh HK, Ali RB, Li BF (October 2001). "The modified human DNA repair enzyme O(6)-methylguanine-DNA methyltransferase is a negative regulator of estrogen receptor-mediated transcription upon alkylation DNA damage". Mol. Cell. Biol. 21 (20): 7105–14. PMC 99886. PMID 11564893. doi:10.1128/MCB.21.20.7105-7114.2001.
Véxase tamén
[editar | editar a fonte]Outros artigos
[editar | editar a fonte]Bibliografía
[editar | editar a fonte]- Margison GP, Povey AC, Kaina B, Santibáñez Koref MF (2003). "Variability and regulation of O6-alkylguanine-DNA alkyltransferase". Carcinogenesis 24 (4): 625–35. PMID 12727789. doi:10.1093/carcin/bgg005.