Proteína AAA
| ATPases associated with diverse cellular activities | |||||||||
|---|---|---|---|---|---|---|---|---|---|
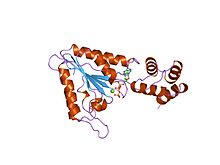 Estrutura do factor sensible á N-etilmaleimida.[1] | |||||||||
| Identificadores | |||||||||
| Símbolo | AAA | ||||||||
| Pfam | PF00004 | ||||||||
| Pfam clan | CL0023 | ||||||||
| InterPro | IPR003959 | ||||||||
| PROSITE | PDOC00572 | ||||||||
| SCOPe | 1nsf / SUPFAM | ||||||||
| CDD | cd00009 | ||||||||
| Membranome | 74 | ||||||||
| |||||||||
As proteínas AAA ou ATPases Asociadas con diversas Actividades celulares son unha familia de proteínas que comparten un módulo conservado común de aproximadamente 230 residuos de aminoácidos. Esta é unha familia proteica grande e funcionalmente diversa que pertence á superfamilia de proteínas AAA+ de NTPases de bucle P con forma de anel, que exercen as súas actividades por medio da remodelación dependente de enerxía ou translocación de moléculas.[2][3]
As proteínas AAA acoplan a enerxía química proporcionada pola hidrólise do ATP a cambios conformacionais que son transducidos a forza mecánica exercida sobre un substrato macromolecular.[4]
As proteínas AAA son diversas en función e organización e varían en actividade, estabilidade e mecanismo.[4] Os membros da familia AAA encóntranse en todos os organismos[5] e son esenciais para moitas funcións celulares. Están implicados en procesos como a replicación do ADN, degradación de proteínas, fusión de membranas, corte de microtúbulos, bioxénese dos peroxisomas, transdución de sinais e regulación da expresión xenética.
Estrutura
[editar | editar a fonte]O dominio AAA contén dous subdominios, un dominio alfa/beta N-terminal que se une a nucleótidos e os hidroliza (un pregamento de Rossmann) e un dominio en hélice alfa C-terminal.[5] O dominio N-terminal ten unha lonxitude de 200-250 aminoácidos e contén motivos Walker A e Walker B,[5] e son compartidos por outras NTPases de bucle P, a superfamilia proteica que inclúe a familia AAA.[6] A maioría das proteínas AAA teñen dominios adicionais que se usan para a oligomerización, unión ao substrato e/ou regulación. Estes dominios poden estar situados en posición N- ou C-terminal no módulo AAA.
Clasificación
[editar | editar a fonte]Algunhas clases de proteínas AAA teñen un dominio N-terminal non de ATPase que é seguido por un ou dous dominios AAA (D1 e D2). Nalgunhas proteínas con dous dominios AAA, ambos os dous están ben conservados evolutivamente (como en Cdc48/p97). Noutros, un deles, que pode ser o dominio D2 (como en Pex1p e Pex6p) ou o domino D1 (como en Sec18p/NSF), está mellor conservado evolutivamente.
Aínda que a familia AAA clásica estaba baseada en motivos, a familia foi ampliada usando información estrutural e agora denomínase AAA+.[5]
Relacións evolutivas
[editar | editar a fonte]As proteínas AAA divídense en sete clados básicos, baseándose en elementos de estrutura secundaria incluídos dentro ou preto do núcleo do pregamento AAA, que se chaman: cargador de abrazadeira (clamp loader), iniciador, clásico, helicase da superfamilia III, HCLR, inserción H2 e inserción PS-II.[4]
Estrutura cuaternaria
[editar | editar a fonte]As ATPases AAA ensámblanse en conxuntos oligoméricos (que adoitan ser homohexámeros) que forman unha estrutura con forma de anel cun poro central. Estas proteínas producen un motor molecular que acopla a unión e hidrólise do ATP a cambios nos estados conformacionais que poden ser propagados pola ensamblaxe para que actúen sobre un substrato diana translocándoo ou remodelándoo.[7]
O poro central pode estar implicado no procesamento do substrato. Na configuración hexamérica, o sitio de unión do ATP está situado na interface entre as subunidades. Despois da unión e hidrólise do ATP, os encimas AAA sofren uns cambios conformacionais nos dominios AAA así como nos dominios N. Estes movementos poden transmitirse á proteína substrato.
Mecanismo molecular
[editar | editar a fonte]A hidrólise do ATP feita polas ATPases AAA propúxose que implica o ataque nucleofílico no fosfato gamma do ATP por parte dunha molécula de auga activada, orixinando o movemento dos subdominios AAA N- e C-terminais un en relación ao outro. Este movemento permite exercer forza mecánica, amplificada por outros dominios ATPase que hai dentro da mesma estrutura oligomérica. Os dominios adicionais da proteína permiten a regulación ou direccionamento da forza cara a diferentes obxectivos.[6]
AAAs procariotas
[editar | editar a fonte]As proteínas AAA non están restrinxidas aos eucariotas. Os procariotas teñen AAAs que combinan proteínas chaperonas con actividade proteolítica, por exemplo no complexo ClpAPS, que media a degradación de proteínas e o recoñecemento en Escherichia coli. O recoñecemento básico de proteínas por AAAs pénsase que ocorre por medio de dominios proteicos non pregados da proteína substrato. En HslU, un ClpX/ClpY bacteriano homólogo da familia HSP100 de proteínas AAA, os subdominios N- e C-terminal móvense un cara ao outro cando os nucleótidos se unen e son hidrolizados. Os dominios terminais están máis afastados no estado libre de nucleótidos e máis próximos no estado unido a ADP. Dese modo a apertura da cavidade central queda afectada.
Funcións
[editar | editar a fonte]As proteínas AAA interveñen na degradación de proteínas, fusión de membranas, replicación do ADN, dinámica dos microtúbulos, transporte intracelular, activación transcricional, re-pregamento de proteínas, desensamblase de complexos proteicos e agregados de proteínas.[5][8]
Movemento molecular
[editar | editar a fonte]As dineínas, unha das tres grandes clases de proteínas motoras, son proteínas AAA, as cales acoplan a súa actividade de ATPase ao movemento molecular ao longo de microtúbulos.[9]
A ATPase de tipo AAA Cdc48p/p97 é quizais a proteína AAA mellor estudada. As proteínas secretoras mal pregadas son exportadcas desde o retículo endoplasmático e degradadas pola vía de degradación asociada ao retículo endoplasmático (ERAD). A membrana non funcional e as proteínas luminais extráense do retículo endoplasmático e son degradadas no citosol por proteasomas. A retrotranslocación e extracción do substrato faise coa axuda do complexo Cdc48p(Ufd1p/Npl4p) no lado citosólico da membrana. No lado citosólico é ubiquitinada polos encimas do retículo endoplasmático E2 e E3 antes da degradación polo proteasoma de 26S.
Envío de proteínas a corpos multivesiculares
[editar | editar a fonte]Os corpos multivesiculares son compartimentos endosómicos que clasifican ou escollen proteínas de membrana ubiquitinadas para incorporalas a vesículas. Este proceso implica a acción secuencial de tres complexos multiproteicos, ESCRT I, II e III (ESCRT significa en inglés endosomal sorting complexes required for transport, é dicir, complexos de clasificación endosómica requiridos para o transporte). A Vps4p é unha ATPase de tipo AAA implicada nesta vía de clasificación MVB. Orixinalmente fora identificada como un mutante de vps (vacuolar protein sorting, proteína de clasificación vacuolar) de ”clase E” e despois viuse que catalizaba a disociación de complexos ESCRT. A Vps4p está ancorada por medio de Vps46p á membrana endosómica. Na ensamblaxe de Vps4p colabora a proteína conservada Vta1p, que regula o seu status de oligomerización e actividade de ATPase.
Outras funcións
[editar | editar a fonte]As proteases AAA usan a enerxía da hidrólise do ATP para translocar unha proteína dentro do proteasoma para a súa degradación.
Proeínas humanas que conteñen este dominio
[editar | editar a fonte]Familia das ATPases AAA (HGNC)
[editar | editar a fonte]- Véxase tamén: HGNC.
AFG3L2; ATAD1; ATAD2; ATAD2B; ATAD3A; ATAD3B; ATAD3C; ATAD5; BCS1L; CHTF18; CLBP; CLPP; CLPX; FIGN; FIGNL1; FIGNL2; IQCA1; KATNA1; KATNAL1; KATNAL2; LONP1; LONP2; MDN1; NSF; NVL; ORC1; ORC4; PEX1; PEX6; PSMC1; PSMC2 (Nbla10058); PSMC3; PSMC4; PSMC5; PSMC6; RFC1; RFC2; RFC3; RFC4; RFC5; RUVBL1; RUVBL2; SPAST; SPATA5 (SPAF); SPATA5L1; SPG7; TRIP13; VCP; VPS4A; VPS4B; WRNIP1; YME1L1 (FTSH);[10]
Torsinas
[editar | editar a fonte]TOR1A; TOR1B; TOR2A; TOR3A; TOR4A;[11]
Outros
[editar | editar a fonte]Pseudoxenes
[editar | editar a fonte]AFG3L1P;[13]
Notas
[editar | editar a fonte]- ↑ Yu RC, Hanson PI, Jahn R, Brünger AT (setembro de 1998). "Structure of the ATP-dependent oligomerization domain of N-ethylmaleimide sensitive factor complexed with ATP". Nat. Struct. Biol. 5 (9): 803–11. PMID 9731775. doi:10.1038/1843.
- ↑ Koonin EV, Aravind L, Leipe DD, Iyer LM (2004). "Evolutionary history and higher order classification of AAA ATPases". J. Struct. Biol. 146 (1–2): 11–31. PMID 15037234. doi:10.1016/j.jsb.2003.10.010.
- ↑ Lupas AN, Frickey T (2004). "Phylogenetic analysis of AAA proteins". J. Struct. Biol. 146 (1–2): 2–10. PMID 15037233. doi:10.1016/j.jsb.2003.11.020.
- ↑ 4,0 4,1 4,2 Erzberger JP, Berger JM (2006). "Evolutionary relationships and structural mechanisms of AAA proteins". Annu. Rev. Biophys. Biomol. Struct. 35: 93–114. PMID 16689629. doi:10.1146/annurev.biophys.35.040405.101933.
- ↑ 5,0 5,1 5,2 5,3 5,4 Hanson PI, Whiteheart SW (xullo de 2005). "AAA proteins: have engine, will work". Nat. Rev. Mol. Cell Biol. 6 (7): 519–29. PMID 16072036. doi:10.1038/nrm1684.
- ↑ 6,0 6,1 Snider J, Thibault G, Houry WA (2008). "The AAA superfamily of functionally diverse proteins". Genome Biol. 9 (4): 216. PMC 2643927. PMID 18466635. doi:10.1186/gb-2008-9-4-216.
- ↑ Smith DM, Benaroudj N, Goldberg A (2006). "Proteasomes and their associated ATPases: A destructive combination". J. Struct. Biol. 156 (1): 72–83. PMID 16919475. doi:10.1016/j.jsb.2006.04.012.
- ↑ Tucker PA, Sallai L (decembro de 2007). "The AAA superfamily--a myriad of motions". Curr. Opin. Struct. Biol. 17 (6): 641–52. PMID 18023171. doi:10.1016/j.sbi.2007.09.012.
- ↑ Carter AP, Vale RD (febreiro de 2010). "Communication between the AAA ring and microtubule-binding domain of dynein". Biochem Cell Biol 88 (1): 15–21. PMC 2894566. PMID 20130675. doi:10.1139/o09-127.
- ↑ "Gene group: AAA ATPases (ATAD)". HUGO Gene Nomenclature Committee.
- ↑ "Gene group: Torsins (TOR)". HUGO Gene Nomenclature Committee.
- ↑ "Symbol report for AK6". HUGO Gene Nomenclature Committee.
- ↑ "Symbol report for AFG3L1P". HUGO Gene Nomenclature Committee.
Véxase tamén
[editar | editar a fonte]Ligazóns externas
[editar | editar a fonte]- Snider J, Houry WA (febreiro de 2008). "AAA proteins: diversity in function, similarity in structure". Biochem. Soc. Trans. 36 (Pt 1): 72–7. PMID 18208389. doi:10.1042/BST0360072.
- White SR, Lauring B (decembro de 2007). "AAA ATPases: achieving diversity of function with conserved machinery". Traffic 8 (12): 1657–67. PMID 17897320. doi:10.1111/j.1600-0854.2007.00642.x.
