Reparación de discordancias no ADN
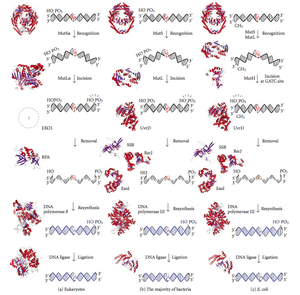
A reparación de discordancias no ADN (abreviada como MMR, do inglés mismatch repair) é un sistema para o recoñecemento e reparación da inserción errada, deleción, ou incorrecta incorporación e bases (sen respectar a complementariedade), que se pode orixinar durante a replicación do ADN e a recombinación, e tamén para a reparación dalgunhas formas de danos no ADN.[1][2]
A reparación de discordancias é específica de febra. Durante a síntese de ADN a febra nova sintetizada (febra filla) incluirá comunmente erros. Para empezar a repararse, a maquinaria de reparación distingue a febra filla de nova síntese da febra molde (parental). En bacterias gramnegativas, a hemimetilación transitoria distingue as dúas febras, xa que a parental está metilada e a filla non. Porén, noutros procariotas e eucariotas, o mecanismo exacto non está claro. Sospéitase que, en eucariotas, a febra retardada do ADN de nova síntese contén transitoriamente amosegas (antes de que sexan seladas pola ADN ligase), o que proporciona un sinal que dirixe os sistemas de corrección de probas de discordancias cara á febra apropiada. Isto implica que estas amosegas deben estar presentes na febra guía, e atopáronse recentemente probas sobre isto.[3] En traballos recentes[4] demostrouse que as amosegas son sitios para a carga dependente de RFC da abrazadeira escorregante de replicación PCNA, dunha maneira específica de orientación, de tal maneira que unha das caras da proteína con forma de donut se xustapón cara ao extremo 3'-OH na amosega. A PCNA orientada dirixe despois a acción da endonuclease MutLalpha cara a unha febra en presenza dunha discordancia e MutSalpha ou MutSbeta.
Calquera evento mutacional que distorsione a estrutura de superhélice do ADN ten o potencial de comprometer a estabilidade xenética da célula. O feito de que os sistemas de reparación e detección de danos sexan tan complexos coma a propia maquinaria de replicación subliña a importancia que na evolución se lle deu á fidelidade na copia do ADN.
Exemplos de bases discordantes son os apareamentos G/T ou A/C, que non respectan o apareamento normal de bases (ver reparación do ADN). As discordancias débense comunmente á tautomerización de bases durante a fase G2 do ciclo celular. Os danos son reparados por recoñecemento da deformidade causada pola discordancia, o que determina as febras molde e non molde, e escinde a base incorporada incorrectamente e substitúea co nucleótido correcto. O proceso de eliminaciónn implica xeralmante a máis nucleótidos que o propio nucleótido discordante. Poden eliminarse xunto con el uns poucos pares de bases ou ás veces ata miles de pares de bases da febra de ADN de nova síntese.
Proteínas da reparación de discordancias
[editar | editar a fonte]| Reparación de discordancias no ADN | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 hpms2-atpgs | |||||||||
| Identificadores | |||||||||
| Símbolo | DNA_mis_repair | ||||||||
| Pfam | PF01119 | ||||||||
| Pfam clan | CL0329 | ||||||||
| InterPro | IPR013507 | ||||||||
| PROSITE | PDOC00057 | ||||||||
| SCOPe | 1bkn / SUPFAM | ||||||||
| |||||||||
A reparación de discordancias é un proceso altamente conservado desde os procariotas aos eucariotas. A primeira proba da reparación de discordancias obtívose na bacteria Streptococcus pneumoniae (xenes hexA e hexB). Traballos posteriores en Escherichia coli identificaron varios xenes que, cando son inactivados mutacionalmente, orixinan cepas hipermutables. Os produtos xénicos denomínanse por esa razón proteínas "Mut", e son os principais compoñentes activos do sistema de reparación de discordancias. Tres destas proteínas son esenciais para detectar a discordancia e dirixir a maquinaria de reparación cara a elas: MutS, MutH e MutL (MutS é un homólogo de HexA e MutL de HexB).
MutS forma un dímero (MutS2) que recoñece a base discordante sobre a febra filla e únese ao ADN mutado. A MutH únese a sitios hemimetilados ao longo da febra de ADN filla, mais a súa acción é latente, e só é activada polo contacto co dímero MutL (MutL2), o cal se une ao complexo MutS-ADN e actúa como un mediador entre MutS2 e MutH, activando este último. O ADN é dobrado en bucle para buscar o sitio de metilación d(GATC) máis próximo á discordancia, o cal pode estar ata a 1 kb de distancia. Despois da activación polo complexo MutS-ADN, a MutH fai unha amosega na febra filla preto do sitio hemimetilado e recruta unha helicase UvrD (ADN helicase II) para separar as dúas febras cunha polaridade específica 3' a 5'. O complexo MutSHL completo deslízase entón ao longo do ADN na dirección da discordancia, liberando a febra para que sexa escindida a medida que avanza. Unha exonuclease rastrea o complexo e dixire a cola de ADN de febra simple. O tipo de exonuclease recrutada depende do lado da discordancia en que MutH cortou a febra, o 5’ ou o 3’. Se a amosega feita por MutH está no extremo 5’ da discordancia, utilízase a exonuclease RecJ ou a ExoVII (ambas as dúas son exonucleases 5’-3’). Porén, se a amosega está no extremo 3’ da discordancia, utilízase a ExoI (un encima 3'-5').
Todo o proceso remata ao superar o sitio da discordancia, é dicir, o propio sitio e os nucleótidos que o rodean son completamente escindidos. O oco de febra simple creado pola exonuclease pode despois ser reparado pola ADN polimerase III (axudada por unha proteína de unión á febra simple), que utiliza a outra febra como molde, e finalmente é selado pola ADN ligase. A ADN metilase despois metila rapidamente a febra filla.
Homólogos de MutS
[editar | editar a fonte]O dímero da MutS2 cando se une ao ADN dobra a hélice do ADN e abrangue aproximadamente uns 20 pares de bases. Ten unha actividade débil de ATPase, e a unión a ela do ATP causa a formación de estruturas terciarias na superficie da molécula. A estrutura cristalina de MutS revela que é excepcionalmente asimétrica, e, aínda que a súa conformación activa é un dímero, só unha das dúas metades interacciona co sitio da discordancia.
En eucariotas, os homólogos de MutS (chamados Msh) forman dous heterodímeros principais: Msh2/Msh6 (MutSα) e Msh2/Msh3 (MutSβ). A vía MutSα está implicada principalmente na substitución de bases e reparacións de discordancias de bucle pequeno. A vía MutSβ tamén está implicada na reparación de bucles pequenos, ademais de na reparación de bucles grandes (bucles de ~10 nucleótidos). Porén, a MutSβ non repara substitucións de bases.
Homólogos de MutL
[editar | editar a fonte]A MutL ten tamén unha débil actividade de ATPase (usa o ATP para impulsar o seu movemento). Forma un complexo con MutS e MutH, o que incrementa a pegada de MutS no ADN.
Porén, a procesividade (a distancia na que o encima pode moverse ao longo do ADN antes de disociarse) de UvrD é de só ~40–50 bp. Como a distancia entre a amosega creada por MutH e discordancia pode ser como media de ~600 pares de bases, se non se cargou outra UvrD a sección non desenrolada do ADN pode despois reenrolarse libremente por apareamento de bases coa súa febra complementaria, forzando o proceso a comezar de novo. Porén, cando é axudado por MutL, a taxa de carga de UvrD increméntase moito. Aínda que a procesividade (e a utilización de ATP) de moléculas individuais de UvrD permanece igual, o efecto total sobre o ADN é pulado considerablemente; o ADN non ten a posibilidade de reenrolarse, xa que cada UvrD desenrola de 40 a 50 pares de bases, disóciase, e despois é inmediatamente substituída por outra UvrD, repetindo o proceso. Isto expón grandes seccións do ADN á dixestión pola exonuclease, permitíndolle unha rápida escisión (e posterior substitución) do ADN incorrecto.
Os eucariotas teñen homólogos de MutL chamados Mlh1 e Pms1. Forman un heterodímero que imita a MutL de E. coli. O homólogo humano da MutL procariota ten tres formas designadas MutLα, MutLβ e MutLγ. O complexo MutLα está formado por dúas subunidades, que son MLH1 e PMS2; o heterodímero MutLβ está constituído por MLH1 e PMS1, mentres que MutLγ está formado por MLH1 e MLH3. A MutLα actúa como facilitador, coordinando os eventos da reparación de discordancias. Descubriuse recentemente que unha ADN endonuclease que introduce roturas de febras no ADN despois da activación por discordancias e por outras proteínas necesarias, MutSa e PCNA. Estas interrupcións nas febras serven como puntos de entrada para a actividade exonuclease que elimina o ADN discordante. Os papeis que xogou MutLβ e MutLγ na reparación de discordancias coñécense menos.
MutH: unha endonuclease presente en E. coli e Salmonella
[editar | editar a fonte]MutH é unha endonuclease moi débil que se activa unha vez que se uniu a MutL (ao cal á súa vez se une a MutS). Fai amosegas no ADN non metilado e na febra non metilada do ADN hemimetilado pero non fai amosegas no ADN completamente metilado. Atopouse en experimentos que a reparación de discordancias é aleatoria se ningunha das febras está metilada. Estes comportamentos fixeron que se propuxese que MutH determina cal das febras contén a discordancia. MutH non ten homólogos en eucariotas. A súa función de endonuclease é realizda neles polos homólogos de MutL, os cales teñen certa actividade especializada de exonuclease 5'-3'. A preferencia para a eliminación das discordancias da febra filla de nova síntese en eucariotas poden proporcionala os extremos 3' libres dos fragmentos de Okazaki da febra de nova síntese durante a replicación.
A PCNA e a abrazadeira escorregante β
[editar | editar a fonte]A PCNA e a abrazadeira escorregante β asócianse con MutSα/β e MutS, respectivamente. Aínda que os primeiros informes suxerían que o complexo PCNA-MutSα pode potenciar o recoñecemento da discordancia,[5] demostrouse recentemente[6] que non hai un cambio aparente na afinidade de MutSα polas discordancias en presenza ou ausencia de PCNA. Ademais, os mutantes para MutSα que non poden interaccionar con PCNA in vitro mostran a capacidade de levar a cabo o recoñecemento e escisión de discordancias a niveis case de tipo salvaxe (normais, non mutantes). Tales mutantes son deficientes na reacción de reparación dirixida por unha rotura de febra 5', o que suxire por primeira vez unha función de MutSα nun paso postescisión da reacción.
Importancia clínica
[editar | editar a fonte]Defectos na reparación de discordancias
[editar | editar a fonte]As mutacións nos homólogos humanos das proteínas Mut afectan á estabilidade xenómica, que pode resultar en inestabilidade de microsatélites. A inestabilidade de microsatélites está implicada na maioría dos cancros humanos. Concretamente, a inmensa maioría dos cancros colorrectais non poliposos hereditarios son atribuídos a mutacións nos xenes que codifican os homólogos de MutS e MutL, que son MSH2 e MLH1 respectivamente, o cal serve para clasificalos como xenes supresores de tumores. Un subtipo deste tipo de cancro colorrectal denomínase síndrome de Muir-Torre, que está asociado con tumores de pel.
A síndrome de cancro de reparación de discordancias (tamén chamada deficiencia na reparación de discordancias ou síndrome de Turcot) combina tipicamente a polipose adenomatosa familiar con tumores cerebrais.[7]
Defectos epixenéticos no cancro
[editar | editar a fonte]Só unha minoría dos cancros esporádicos cunha deficiencia de reparación do ADN teñen unha mutación nun xene de reparación do ADN. Porén, a maioría dos cancros esporádicos cunha deficiencia na reparación do ADN teñen unha ou máis alteracións epixenéticas que reducen ou silencian a expresión dos xenes de reparación de ADN.[8] Un 13% dos cancros colorrectais son deficientes na reparación das discordancias no ADN, e un 9,8% debido a unha perda da MLH1, e porcentaxes menores debido á perda da MSH2 (1,4%), MSH6 (0,5%) e PMS2 (1,5%).[9] En 65 de 66 casos de cancros esporádicos nos cales MLH1 era deficiente, a deficiencia era debida á metilación da rexión promotora do xene MLH1.[9]
Outros cancros teñen maiores frecuencias de perda da reparación de discordancias debido á perda da MLH1 (ver táboa abaixo). Como se indica na táboa, avaliáronse os defectos na reparación de discordancias que se debían á perda da expresión de MLH1, e encontrouse que as deficiencias da MLH1 eran en gran medida o resultado da metilación da rexión promotora do xene MLH1.
Outro mecanismo esporádico polo cal se reducen as expresións da MLH1 e MSH2 é a sobreexpresión de miR-155.[10] MiR-155 ten como diana a MLH1 e MSH2. Nos cancros humanos colorrectais atopouse unha correlación inversa entre a expresión de miR-155 e a expresión das proteínas MLH1 ou MSH2.[10]
Expresión deficiente en cancros
[editar | editar a fonte]| Tipo de cancro | Frecuencia da deficiencia en cancros | Frecuencia da deficiencia en defectos de campo adxacentes |
|---|---|---|
| Estómago | 32%[11][12] | 24%-28% |
| Estómago (tumores de tipo foveolar) | 74%[13] | 71% |
| Estómago con alta incidencia no val de Caxemira | 73%[14] | 20% |
| Esófago | 73%[15] | 27% |
| Carcinoma de células escamosas de cabeza e pescozo | 31%-33%[16][17] | 20%-25% |
| Cancro de pulmón de células non pequenas | 69%[18] | 72% |
| Colorrectal | 10%[9] |
Deficiencia nos defectos de campo
[editar | editar a fonte]Un defecto de campo é unha área ou "campo" de epitelio que foi precondicionado por cambios epixenéticos e/ou mutacións, polo que predispón ao desenvolvemento de cancro. Como indicou Rubin, "A gran maioría dos estudos na investigación do cancro fixéronse en tumores ben definidos in vivo, ou en focos neoplásticos discretos in vitro.[19] Porén, hai probas de que máis do 80% das mutacións somáticas encontradas en fenotipos mutadores humanos colorrectais ocorren antes do comezo da expansión clonal terminal."[20] De xeito similar, Vogelstein et al.[21] sinalaron que máis da metade das mutacións somáticas identificadas en tumores ocorrían na fase preneoplástica (nun defecto de campo), durante o crecemento de células aparentemente normais.
Na táboa de arriba, detectáronse deficiencias en MLH1 nos defectos de campo (tecidos histoloxicamente normais) que rodeaban a maioría dos cancros. Se a MLH1 é reducida epixeneticamente ou silenciada, probablemente non lle confire vantaxe selectiva a unha célula nai. Porén, a expresión reducida ou ausente da MLH1 causaría un incremento das taxas de mutación, e un ou máis dos xenes mutados poden proporcionar á célula vantaxes selectivas. O xene MLH1 con expresión deficiente podería entón ser portado como xene un pasaxeiro (autostopista) selectivamente neutro ou só lixeiramente deletéreo cando a célula nai mutada xera un clon expandido. A presenza continua dun clon cun MLH1 reprimido epixeneticamente continuaría xerando máis mutacións, algunhas das cales poderían producir un tumor.
Deficiencia da MMR en humanos
[editar | editar a fonte]En humanos, sete proteínas de repaación de discordancias no ADN (MLH1, MLH3, MSH2, MSH3, MSH6, PMS1 e PMS2) traballan coordinaamente en pasos secuenciais para iniciar a reparación das discordancias.[22] Ademais, hai subvías dependentes de Exo1 e independentes de Exo1.[23]
Outros produtos xénicos implicados na reparación de discordancias (despois de feita a iniciación por xenes da reparación de discordancias) en humanos inclúen a ADN polimerase delta, a PCNA, a RPA, a HMGB1, o RFC e a ADN ligase I, xunto con factores modificadores das histonas e a cromatina.[24][25]
Carencia de MMR e frecuencia de mutación
[editar | editar a fonte]O recoñecemento e reparación de discordancias son importantes para as células porque se non se fan o resultado é inestabilidade de microsatélites e unha elevada taxa de mutación espontánea (fenotipo mutador). Entre 20 cancros avaliados o cancro de colon inestable en microsatálites deficiente na reparación de discordancias tiña a segunda frecuencia de mutación máis alta (despois do melanoma).
Porén, a ausencia de reparación de discordancias a miúdo ocorre en coordinación coa perda doutros xenes de reparación do ADN.[8] Nun exemplo, que implica aos xenes MLH1 e MLH3, Jiang et al.[26] realizaron un estudo no que avaliaron a expresión dos ARNm de 27 xenes de reparación do ADN en 40 astrocitomas comparada coa de tecidos cerebrais normais de individuos sen astrocitoma. Entre os 27 xenes de reparación do ADN avaliados, 13 xenes de reparación do ADN, que eran MLH1, MLH3, MGMT, NTHL1, OGG1, SMUG1, ERCC1, ERCC2, ERCC3, ERCC4, RAD50, XRCC4 e XRCC5, estaban todos significativamente regulados á baixa en cada un dos tres graos (II, III e IV) dos astrocitomas. A represión destes 13 xenes en astrocitomas de grao inferior e tamén de alto grao suxeriu que poderían ser importantes nos estadios iniciais e nos avanzados dos astrocitomas. Noutro exemplo, Kitajima et al.[27] encontraron que a inmunorreactividade en 135 espécimes de cancro gástrico e a perda da expresión de MLH1 e MGMT estaba estreitamente correlacionada e parecían estar sincronicamente aceleradas durante a progresión do tumor.
A expresión deficiente de moitos xenes de reparación do ADN atópase a miúdo en cancros,[8] e pode contribuír á aparición de miles de mutacións que se encontran usualmente en cancros.
Notas
[editar | editar a fonte]- ↑ Iyer R, Pluciennik A, Burdett V, Modrich P (2006). "DNA mismatch repair: functions and mechanisms". Chem Rev 106 (2): 302–23. PMID 16464007. doi:10.1021/cr0404794.
- ↑ Larrea AA, Lujan SA, Kunkel TA (2010). "DNA mismatch repair". Cell 141 (4): 730. PMID 20478261. doi:10.1016/j.cell.2010.05.002.
- ↑ Heller RC, Marians KJ (2006). "Replisome assembly and the direct restart of stalled replication forks". Nat Rev Mol Cell Biol 7 (12): 932–43. PMID 17139333. doi:10.1038/nrm2058.
- ↑ Pluciennik; et al. (2010). "PCNA function in the activation and strand direction of MutLα endonuclease in mismatch repair.". PNAS 107 (37): 16066–71. PMC 2941292. PMID 20713735. doi:10.1073/pnas.1010662107.
- ↑ Flores-Rozas H, Clark D, Kolodner RD (2000). "Proliferating cell nuclear antigen and Msh2p-Msh6p interact to form an active mispair recognition complex". Nature Genetics 26 (3): 375–8. PMID 11062484. doi:10.1038/81708.
- ↑ Iyer RR, Pohlhaus TJ, Chen S, Hura GL, Dzantiev L, Beese LS, Modrich P (2008). "The MutSalpha-proliferating cell nuclear antigen interaction in human DNA mismatch repair". Journal of Biological Chemistry 283 (19): 13310–9. PMC 2423938. PMID 18326858. doi:10.1074/jbc.M800606200.
- ↑ Online 'Mendelian Inheritance in Man' (OMIM) 276300
- ↑ 8,0 8,1 8,2 Bernstein C, Bernstein H (2015). "Epigenetic reduction of DNA repair in progression to gastrointestinal cancer". World J Gastrointest Oncol 7 (5): 30–46. PMC 4434036. PMID 25987950. doi:10.4251/wjgo.v7.i5.30.
- ↑ 9,0 9,1 9,2 Truninger K, Menigatti M, Luz J, Russell A, Haider R, Gebbers JO, Bannwart F, Yurtsever H, Neuweiler J, Riehle HM, Cattaruzza MS, Heinimann K, Schär P, Jiricny J, Marra G (2005). "Immunohistochemical analysis reveals high frequency of PMS2 defects in colorectal cancer". Gastroenterology 128 (5): 1160–71. PMID 15887099. doi:10.1053/j.gastro.2005.01.056.
- ↑ 10,0 10,1 Valeri N, Gasparini P, Fabbri M, Braconi C, Veronese A, Lovat F, Adair B, Vannini I, Fanini F, Bottoni A, Costinean S, Sandhu SK, Nuovo GJ, Alder H, Gafa R, Calore F, Ferracin M, Lanza G, Volinia S, Negrini M, McIlhatton MA, Amadori D, Fishel R, Croce CM (2010). "Modulation of mismatch repair and genomic stability by miR-155". Proc. Natl. Acad. Sci. U.S.A. 107 (15): 6982–7. PMC 2872463. PMID 20351277. doi:10.1073/pnas.1002472107.
- ↑ Kupčinskaitė-Noreikienė R, Skiecevičienė J, Jonaitis L, Ugenskienė R, Kupčinskas J, Markelis R, Baltrėnas V, Sakavičius L, Semakina I, Grižas S, Juozaitytė E (2013). "CpG island methylation of the MLH1, MGMT, DAPK, and CASP8 genes in cancerous and adjacent noncancerous stomach tissues". Medicina (Kaunas) 49 (8): 361–6. PMID 24509146.
- ↑ Waki T, Tamura G, Tsuchiya T, Sato K, Nishizuka S, Motoyama T (2002). "Promoter methylation status of E-cadherin, hMLH1, and p16 genes in nonneoplastic gastric epithelia". Am. J. Pathol. 161 (2): 399–403. PMC 1850716. PMID 12163364. doi:10.1016/S0002-9440(10)64195-8.
- ↑ Endoh Y, Tamura G, Ajioka Y, Watanabe H, Motoyama T (2000). "Frequent hypermethylation of the hMLH1 gene promoter in differentiated-type tumors of the stomach with the gastric foveolar phenotype". Am. J. Pathol. 157 (3): 717–22. PMC 1949419. PMID 10980110. doi:10.1016/S0002-9440(10)64584-1.
- ↑ Wani M, Afroze D, Makhdoomi M, Hamid I, Wani B, Bhat G, Wani R, Wani K (2012). "Promoter methylation status of DNA repair gene (hMLH1) in gastric carcinoma patients of the Kashmir valley". Asian Pac. J. Cancer Prev. 13 (8): 4177–81. PMID 23098428. doi:10.7314/apjcp.2012.13.8.4177.
- ↑ Chang Z, Zhang W, Chang Z, Song M, Qin Y, Chang F, Guo H, Wei Q (2015). "Expression characteristics of FHIT, p53, BRCA2 and MLH1 in families with a history of oesophageal cancer in a region with a high incidence of oesophageal cancer". Oncol Lett 9 (1): 430–436. PMC 4246613. PMID 25436004. doi:10.3892/ol.2014.2682.
- ↑ Tawfik HM, El-Maqsoud NM, Hak BH, El-Sherbiny YM (2011). "Head and neck squamous cell carcinoma: mismatch repair immunohistochemistry and promoter hypermethylation of hMLH1 gene". Am J Otolaryngol 32 (6): 528–36. PMID 21353335. doi:10.1016/j.amjoto.2010.11.005.
- ↑ Zuo C, Zhang H, Spencer HJ, Vural E, Suen JY, Schichman SA, Smoller BR, Kokoska MS, Fan CY (2009). "Increased microsatellite instability and epigenetic inactivation of the hMLH1 gene in head and neck squamous cell carcinoma". Otolaryngol Head Neck Surg 141 (4): 484–90. PMID 19786217. doi:10.1016/j.otohns.2009.07.007.
- ↑ Safar AM, Spencer H, Su X, Coffey M, Cooney CA, Ratnasinghe LD, Hutchins LF, Fan CY (2005). "Methylation profiling of archived non-small cell lung cancer: a promising prognostic system". Clin. Cancer Res. 11 (12): 4400–5. PMID 15958624. doi:10.1158/1078-0432.CCR-04-2378.
- ↑ Rubin H (March 2011). "Fields and field cancerization: the preneoplastic origins of cancer: asymptomatic hyperplastic fields are precursors of neoplasia, and their progression to tumors can be tracked by saturation density in culture". BioEssays 33 (3): 224–31. PMID 21254148. doi:10.1002/bies.201000067.
- ↑ Tsao JL, Yatabe Y, Salovaara R, Järvinen HJ, Mecklin JP, Aaltonen LA, Tavaré S, Shibata D (February 2000). "Genetic reconstruction of individual colorectal tumor histories". Proc. Natl. Acad. Sci. U.S.A. 97 (3): 1236–41. PMC 15581. PMID 10655514. doi:10.1073/pnas.97.3.1236.
- ↑ Vogelstein B, Papadopoulos N, Velculescu VE, Zhou S, Diaz LA, Kinzler KW (March 2013). "Cancer genome landscapes". Science 339 (6127): 1546–58. PMC 3749880. PMID 23539594. doi:10.1126/science.1235122.
- ↑ Pal T, Permuth-Wey J, Sellers TA (2008). "A review of the clinical relevance of mismatch-repair deficiency in ovarian cancer". Cancer 113 (4): 733–42. PMC 2644411. PMID 18543306. doi:10.1002/cncr.23601.
- ↑ Goellner EM, Putnam CD, Kolodner RD (2015). "Exonuclease 1-dependent and independent mismatch repair". DNA Repair (Amst.) 32: 24–32. PMID 25956862. doi:10.1016/j.dnarep.2015.04.010.
- ↑ Li GM (2008). "Mechanisms and functions of DNA mismatch repair". Cell Res. 18 (1): 85–98. PMID 18157157. doi:10.1038/cr.2007.115.
- ↑ Li GM (2014). "New insights and challenges in mismatch repair: getting over the chromatin hurdle". DNA Repair (Amst.) 19: 48–54. PMC 4127414. PMID 24767944. doi:10.1016/j.dnarep.2014.03.027.
- ↑ Jiang Z, Hu J, Li X, Jiang Y, Zhou W, Lu D (2006). "Expression analyses of 27 DNA repair genes in astrocytoma by TaqMan low-density array". Neurosci. Lett. 409 (2): 112–7. PMID 17034947. doi:10.1016/j.neulet.2006.09.038.
- ↑ Kitajima Y, Miyazaki K, Matsukura S, Tanaka M, Sekiguchi M (2003). "Loss of expression of DNA repair enzymes MGMT, hMLH1, and hMSH2 during tumor progression in gastric cancer". Gastric Cancer 6 (2): 86–95. PMID 12861399. doi:10.1007/s10120-003-0213-z.
Véxase tamén
[editar | editar a fonte]Outros artigos
[editar | editar a fonte]Bibliografía
[editar | editar a fonte]- Hsieh P, Yamane K (2008). "DNA mismatch repair: molecular mechanism, cancer, and ageing". Mechanisms of Ageing and Development 129 (7-8): 391–407. PMC 2574955. PMID 18406444. doi:10.1016/j.mad.2008.02.012.
- Iyer RR, Pluciennik A, Burdett V, Modrich PL (Feb 2006). "DNA mismatch repair: functions and mechanisms". Chemical Reviews 106 (2): 302–23. PMID 16464007. doi:10.1021/cr0404794.
- Joseph N, Duppatla V, Rao DN (2006). "Prokaryotic DNA mismatch repair". Progress in Nucleic Acid Research and Molecular Biology 81: 1–49. PMID 16891168. doi:10.1016/S0079-6603(06)81001-9.
- Yang W (Aug 2000). "Structure and function of mismatch repair proteins". Mutation Research 460 (3-4): 245–56. PMID 10946232. doi:10.1016/s0921-8777(00)00030-6.
- Griffiths AJ, Gilbert WM, Lewontin RC, Wessler SR, Suzuki DT, Miller JH (2004). An introduction to genetic analysis (8th ed.). New York, NY: Freeman. ISBN 978-0-7167-4939-4.
- Kunkel TA, Erie DA (2005). "DNA mismatch repair". Annu. Rev. Biochem. 74: 681–710. PMID 15952900. doi:10.1146/annurev.biochem.74.082803.133243.
- Friedberg EC, Walker GC, Siede W, Wood RD, Schultz RA, Ellenberger T (2005). DNA repair and mutagenesis (2nd ed.). Washington, D.C.: ASM Press. ISBN 978-1-55581-319-2.
Ligazóns externas
[editar | editar a fonte]- Reparación do ADN Arquivado 12 de febreiro de 2018 en Wayback Machine.
- DNA Mismatch Repair Medical Subject Headings (MeSH) na Biblioteca Nacional de Medicina dos EUA.
