Metabolismo
O metabolismo (do grego μεταβολή, metabolē, "cambio" ou de μεταβολισμός, metabolismos, "derrocamento") é o conxunto integrado das reaccións químicas que teñen lugar nunha célula ou organismo. Está constítuido por series de reaccións que forman unha rede complexa, ordenada e interconectada de vías ou rutas metabólicas catalizadas por encimas. A función destas reaccións é obter e transformar materia e enerxía. Estas reaccións catalizadas por encimas permiten que os organismos crezan e se reproduzan, manteñan as súas estruturas, e respondan ao seu medio ambiente. A palabra metabolismo tamén se refire a todas as reaccións químicas que teñen lugar nun organismo vivo, incluíndo a dixestión e o transporte de substancias ás células e entre as distintas células, polo que as reaccións que teñen lugar nas células denomínanse especificamente metabolismo intermediario.
O metabolismo divídese xeralmente en dous conxuntos de reaccións, as que forman o catabolismo e as do anabolismo. Anabolismo e catabolismo son procesos interconectados e simultáneos. O equilibrio entre ambos mantense grazas á regulación do metabolismo. Caracterízanse polo seguinte:
- A parte do metabolismo na que as macromoléculas son oxidadas e degradadas noutras máis pequenas obtendo enerxía é o catabolismo. A enerxía almacénase en forma de ATP. Nas reaccións catabólicas fórmanse coencimas con capacidade redutora. As rutas catabólicas desembocan en rutas centrais, polo que o catabolismo é un proceso converxente.
- A parte que se encarga de construír moléculas grandes a partir doutras máis pequenas (gastando así enerxía e coencimas redutores) é o anabolismo. O anabolismo é un proceso redutor e as súas rutas son diverxentes (non desembocan en rutas centrais).
As reaccións químicas do metabolismo están organizadas en vías metabólicas, nas cales unha substancia química se transforma por medio dunha serie de pasos noutra distinta, pola acción dunha secuencia de encimas. Os encimas son fundamentais no metabolismo porque permiten aos organismos impulsar as reaccións desexables que requiren enerxía e que non ocorren espontaneamente, ao acoplalas a reaccións espontáneas que liberan enerxía. Como os encimas actúan como catalizadores fan que estas reaccións se produzan a gran velocidade e eficientemente. Os encimas tamén permiten a regulación das vías metabólicas en resposta a cambios no ambiente da célula ou por causa de sinais procedentes doutras células.
O metabolismo dun organismo determina que substancias son para el nutritivas e cales son velenosas. Por exemplo, algúns procariotas utilizan o sulfuro de hidróxeno como nutriente, pero este gas é velenoso para os animais.[1] A velocidade do metabolismo, chamada taxa metabólica, inflúe na cantidade de alimento que require un organismo, e tamén afecta a como pode obtelo.
Unha característica notable do metabolismo é a semelanza de todas as vías metabólicas básicas e dos seus compoñentes entre as máis diversas especies.[2] Por exemplo, o conxunto de ácidos carboxílicos que funcionan como intermediarios do ciclo do ácido cítrico está presente en todos os organismos coñecidos, e encóntrase en especies tan diversas como organismos unicelulares como a bacteria Escherichia coli e organismos pluricelulares enormes como os elefantes ou as baleas.[3] Estas rechamantes semellanzas nas vías metabólicas débense probablemente á súa aparición en época moi temperá na historia evolutiva da vida, e a que foron conservadas debido á súa grande eficacia.[4][5]
Tipos de metabolismo
[editar | editar a fonte]Os tipos de metabolismos poden clasificarse de diversas maneiras:
- Segundo o modo de obter enerxía os organismos poden clasificarse en:
- Quimiótrofos: obteñen a enerxía de reaccións químicas entre as súas moléculas durante as oxidacións do catabolismo por medio da respiración celular ou da fermentación. Na respiración poden oxidar substancias orgánicas (o máis común) ou inorgánicas (só certas bacterias).
- Fotótrofos: obteñen a enerxía da luz do sol por medio da fotosíntese. Case todos os fotótrofos son autótrofos (fotoautótrofos), pero hai unhas poucas especies de bacterias fotoheterótrofas.
- Segundo a fonte de carbono utilizada poden ser:
- Heterótrofos: utilizan carbono procedente de substancias orgánicas, que foron fabricadas por outros seres vivos.
- Autótrofos: utilizan carbono de procedencia inorgánica, do CO2.
- Segundo a fonte de electróns e hidróxenos usada para obter enerxía poden ser:
- Organótrofos: a súa fonte de electróns son as substancias orgánicas e obteñen a enerxía por medio de reaccións entre substancias orgánicas.
- Litótrofos:[6] a súa fonte de electróns son substancias inorgánicas e obteñen a enerxía de reaccións entre substancias inorgánicas. Os seus representantes máis comúns son as bacterias litoautótrofas, que respiran (oxidan) substancias inorgánicas e que toman o carbono do CO2, polo que non necesitan moléculas orgánicas nin para producir enerxía nin para obter carbono.
- Segundo o seu uso de oxíxeno poden ser:
- Aerobios: Necesitan oxíxeno para vivir. O proceso aerobio básico é a respiración aerobia. O oxíxeno funciona como aceptor final de electróns na cadea de transporte de electróns da respiración celular aerobia. Os microaerófilos son aerobios pero crecen mellor a un nivel moi baixo de oxíxeno (a niveis máis altos certos encimas que posúen quedan inactivados).
- Anaerobios: Non necesitan oxíxeno para vivir. Procesos que non utilizan oxíxeno son a fermentación e a respiración anaerobia (esta non utiliza o oxíxeno como aceptor final de electróns, senón outro composto). Poden ser anaerobios estritos (ou obrigados) se para eles o oxíxeno é letal. Son anaerobios facultativos se poden vivir con ou sen oxíxeno, cambiando o seu metabolismo segundo a presenza de oxíxeno no seu medio (pasando da respiracion aerobia á fermentación, por exemplo). Os aerotolerantes non usan o oxíxeno pero poden tolerar certas concentracións de oxíxeno.
A maior diversidade de metabolismos atópase entre os procariotas, entre os que atopamos todos os tipos de metabolismo dependendo das especies, e mesmo algunhas especies son moi versátiles e poden cambiar de metabolismo segundo as circunstancias.
As biomoléculas do metabolismo
[editar | editar a fonte]- Artigos principais: Biomolécula e Macromolécula.
A maior parte das estruturas que compoñen os seres vivos están formadas por proteínas, carbohidratos, lípidos e ácidos nucleicos. Estas moléculas están formadas por outras máis pequenas que se unen formando os polímeros e outras macromoléculas biolóxicas. Entre estas moléculas básicas están os aminoácidos, monosacáridos, nucleótidos e ácidos graxos. Como estas moléculas son vitais para a vida, o metabolismo céntrase en sintetizalas a partir doutras moléculas máis pequenas e fáciles de obter, pero algunhas non poden ser sintetizadas polo metabolismo de moitos animais, polo que se deben obter da dieta, como ocorre cos aminoácidos esenciais, os ácidos graxos esenciais e as vitaminas, pero outros seres vivos si poden sintetizalas. Algunhas moléculas utilízanse para obter enerxía a partir delas, outras teñen unha función estrutural ou plástica, outras son moléculas catalíticas ou reguladoras. As moléculas con función enerxética son destruídas durante a súa degradación; as outras son en xeral recicladas. As moléculas catalizadoras e reguladoras, como os encimas, coencimas e vitaminas son moi importantes para o funcionamento do metabolismo. Os coencimas transportan grupos químicos entre substratos colaborando cos encimas, e adoitan ter unha porción da súa molécula derivada dalgunha vitamina. Moitas reaccións do metabolismo serían imposibles sen a presenza destes coencimas e vitaminas. Ademais, no metabolismo son moi importantes as substancias inorgánicas do noso corpo, como a auga e os sales minerais. Todo o metabolismo ten lugar en disolución acuosa e a auga pode intervir directamente nas reaccións de hidrólise. Os sales minerais forman ións que interveñen na catálise encimática, entre outras funcións.[7][8][9][10]
Na seguinte táboa móstranse os biopolímeros máis comúns:
| Tipo de molécula | Nome do monómero | Nome do polímero | Exemplos |
|---|---|---|---|
| Proteínas | Aminoácidos | Polipéptidos | proteínas globulares e filamentosas |
| Carbohidratos | Monosacáridos | Polisacáridos | amidón, glicóxeno, celulosa |
| Ácidos nucleicos | Nucleótidos | Polinucleótidos | ADN e ARN |
Coencimas
[editar | editar a fonte]- Artigo principal: Coencima.

Mención especial merecen os coencimas. O metabolismo supón un gran número de reaccións químicas, pero a gran maioría presenta algún dos mecanismos de catálise básicos de reacción de transferencia en grupo.[11] Esta química común permite que as células utilicen unha pequena colección de intermediarios metabólicos para trasladar grupos químicos funcionais entre diferentes reaccións.[9] Estes intermediarios de transferencia de grupos denomínanse coencimas. Cada clase de reacción de grupo lévaa a cabo un coencima en particular, que é o substrato para un grupo de encimas que o producen, e un grupo de encimas que o utilizan. Estes coencimas están a ser decote creados, consumidos e logo reciclados.[12]
O coencima máis importante é a adenosina trifosfato (ATP). Este nucleótido é utilizado para transferir enerxía química entre distintas reaccións químicas. Tamén é útil para transportar grupos fosfato en reacciones de fosforilación. Outro coencima importante é o NADH/NAD+, que intervén en oxidacións-reducións.
Almacenamento de enerxía e poder redutor no metabolismo
[editar | editar a fonte]Os seres vivos obteñen enerxía da oxidación de moléculas e da captación da enerxía do sol, fundamentalmente. Nas oxidacións de moléculas a enerxía libérase en parte en forma de calor e en parte como enerxía libre. A enerxía libre pode almacenarse no coencima adenosina trifosfato (ATP), que é un nucleótido. Nas reaccións nas que se desprende enerxía prodúcese acopladamente a reacción entre o ADP e un fosfato para formar o ATP, onde queda almacenada a enerxía. Despois, o ATP pode soltar o seu último fosfato cedendo enerxía para impulsar reaccións que non son espontáneas ou outros procesos celulares. Polo tanto, o ATP actúa como unha conexión entre o catabolismo e o anabolismo, con reaccións catabólicas que xeran ATP e reaccións anabólicas que o consomen. Ademais do ATP, outros nucleótidos como o GTP, CTP, UTP e TTP, poden facer ese mesmo papel en certas reaccións especializadas, pero non teñen o papel xeral do ATP en todo o metabolismo. A formación de ATP pode realizarse nas propias reaccións por medio de fosforilación a nivel de substrato ou por medio do encima ATP sintase durante as fases finais da respiración celular (fosforilación oxidativa) e da fase luminosa da fotosíntese (fotofosforilación). Só hai unha pequena parte de ATP nas células, pero como é continuamente rexenerado, o corpo humano pode chegar a utilizar o seu propio peso en ATP por día.[12] Outras moléculas fosfatadas ou con xofre teñen enlaces con configuracións electrónicas de alta enerxía, que tamén poden servir para impulsar determinadas reaccións. A nerxía da luz captada polas clorofilas tamén dá lugar, por medio dun mecanismo parecido ao da respiración, á produción de ATP (e de poder redutor).
Outras moléculas necesarias para o metabolismo, fundamentalmente para as reaccións anabólicas, son as que teñen gran capacidade redutora, o que se chama poder redutor. Estas moléculas con poder redutor son fundamentalmente os coencimas que interveñen nas deshidroxenacións de moléculas no metabolismo, xa que ceden facilmente a outras moléculas hidróxenos e electróns. Centos de encimas deshidroxenases eliminan electróns dos seus substratos e reducen o NAD+ a NADH (ou os outros coencimas).[13] Os coencimas con poder redutor máis importantes son o NADH e NADPH (que ceden un hidróxeno e un electrón e se forman no catabolismo ou na fotosíntese), e o FADH2 e FMNH2 (que ceden dous hidróxenos e se forman no catabolismo). Estas moléculas son gastadas no anabolismo e na fase escura da fotosíntese, dando lugar ás súas formas oxidadas NAD+, NADP+, FAD e FMN.
Catabolismo
[editar | editar a fonte]- Artigo principal: Catabolismo.
O catabolismo é o conxunto de procesos metabólicos que degradan as moléculas para liberar enerxía e obter outras moléculas máis simples. No catabolismo degrádanse as moléculas grandes noutras máis pequenas. Estes procesos inclúen a degradación e oxidación das moléculas dos alimentos, e as reaccións que reteñen a enerxía do Sol. O propósito destas reaccións catabólicas é fornecer enerxía, moléculas con capacidade redutora (poder redutor) e compoñentes que se necesitan nas reaccións anabólicas. A natureza destas reaccións catabólicas difire dunha especie a outra. Porén, estas diferentes formas de catabolismo dependen de reaccións de oxidación-redución que implican a transferencia de electróns desde moléculas doantes (como as moléculas orgánicas, auga, amoníaco, sulfuro de hidróxeno e ións de ferro), a aceptores de ditos electróns como o oxíxeno, o nitrato ou o sulfato.[14]
Nos animais, estas reaccións supoñen a degradación de moléculas orgánicas complexas a outras máis simples, como dióxido de carbono e auga. En organismos fotosintéticos como plantas e cianobacterias úsanse as transferencias de electróns como un medio para almacenar enerxía solar.[15]
| Fonte de enerxía | luz solar | fotó- | -trofo | ||
| Moléculas preformadas | quimió- | ||||
| Doante de electróns | composto orgánico | organó- | |||
| composto inorgánico | litó- | ||||
| Fonte de carbono | composto orgánico | heteró- | |||
| composto inorgánico | autó- | ||||
O conxunto máis común de reaccións catabólicas en animais pode ser dividido en tres fases. Na primeira, as moléculas orgánicas grandes como as proteínas, polisacáridos ou lípidos son dixeridas fóra das células orixinando compoñentes máis pequenos. Logo, estas moléculas pequenas son levadas ás células e convertidas en moléculas aínda máis pequenas, xeralmente acetilos que se unen covalentemente ao coencima A, para formar o acetil-coencima A, á vez que se libera enerxía. Finalmente, o grupo acetil do acetil-CoA é oxidado a auga e dióxido de carbono, liberando enerxía que se almacena ao reducir o coencima nicotinamida adenina dinucleótido (NAD+) a NADH.
Dixestión
[editar | editar a fonte]- Artigos principais: Dixestión e Aparato dixestivo.
As células non poden utilizar directamente as macromoléculas como o amidón, a celulosa ou as proteínas, polo que necesitan que se degraden primeiro en unidades máis simples que se poidan usar no metabolismo celular. Hai moitos encimas que dixiren estes polímeros. Entre estes encimas están a peptidase que dixire proteínas a aminoácidos, glicosil hidrolases que dixiren polisacáridos a disacáridos e monosacáridos, e lipases que dixieren os triglicéridos a ácidos graxos e glicerol.
Os microbios simplemente segregan encimas dixestivos fóra da célula[16][17] mentres que os animais segregan estes encimas desde células especializadas á luz do aparato dixestivo.[18] Os aminoácidos, monosacáridos, e triglicéridos liberados por estes encimas extracelulares son absorbidos polas células por medio de proteínas específicas de transporte.[19][20]

Enerxía dos compostos orgánicos
[editar | editar a fonte]O catabolismo de carbohidratos é a degradación destes en unidades menores. Os carbohidratos son xeralmente captados pola célula unha vez que foron dixeridos a monosacáridos.[21] Unha vez dentro da célula, a ruta de degradación utilizada é a glicólise, na cal os azucres como a glicosa e a frutosa son transformados en piruvato á vez que se xeran algunhas moléculas de ATP.[22] O piruvato é un intermediario de varias rutas metabólicas, pero a maioría é convertido en acetil-CoA, que entra no ciclo de Krebs. Aínda que no ciclo se xera máis ATP, o produto máis importante é o NADH, sintetizado a partir do NAD+ pola oxidación do acetil-CoA. A oxidación libera dióxido de carbono como produto de refugallo. Unha ruta alternativa para a degradación da glicosa é a ruta da pentosa fosfato, que reduce o coencima NADPH e produce azucres de 5 carbonos como a ribosa, o azucre que forma parte dos ácidos ribonucleicos.
As graxas son catalizadas por hidrólise a ácidos graxos e glicerol. O glicerol entra na glicólise e os ácidos graxos son degradados por beta-oxidación para liberaren acetil-CoA, que é logo cedido ao ciclo de Krebs. Os ácidos graxos producen máis enerxía que os carbohidratos porque conteñen menos oxíxeno.
Os aminoácidos utilizanse principalmente para sintetizar proteínas e outras biomoléculas; só se oxidan os excedentes orixinando urea e dióxido de carbono como fonte de enerxía.[23] Esta ruta oxidativa empeza coa eliminación do grupo amino por unha aminotransferase. O grupo amino cédese ao ciclo da urea, deixando un esqueleto carbonado en forma de cetoácido.[24] Os aminoácidos glicoxénicos poden ser transformados en glicosa por medio da gliconeoxénese.[25]
- Véxase tamén: Respiración celular e Fermentación.
Fosforilación oxidativa
[editar | editar a fonte]- Artigo principal: Fosforilación oxidativa.
Na fosforilación oxidativa, os electróns liberados das moléculas dos alimento en rutas como o ciclo de Krebs son transferidos ao oxíxeno, e a enerxía é liberada para sintetizar adenosina trifosfato. Isto fano nas células eucariotas unha serie de proteínas situadas nas membranas das mitocondrias que forman a chamada cadea de transporte de electróns. Nas células procariotas, estas proteínas encóntranse na membrana plasmática.[26] Estas proteínas utilizan a enerxía liberada polo paso de electróns desde moléculas reducidas como o coencima NADH ata o oxíxeno para bombear protóns a través da membrana.[27]
Os protóns bombeados fóra da mitocondria crean unha diferenza de concentración a través da membrana, o que xera un gradiente electroquímico.[28] Esta forza fai que volvan á mitocondria a través da subunidade Fo da ATP sintase. O fluxo de protóns fai que parte do encima xire, o que fai que no sitio activo se produza a fosforilación da adenosina difosfato (ADP) e se converta en ATP.[12]
Enerxía dos compostos inorgánicos
[editar | editar a fonte]- Véxase tamén: Ciclo do nitróxeno.
Os procariotas quimiolitótrofos posúen un tipo de metabolismo no que a enerxía se obtén a partir dun composto inorgánico. Estes organismos utilizan hidróxeno,[29] compostos do xofre reducidos (como o sulfuro, sulfuro de hidróxeno e tiosulfato),[1] ión ferroso[30] ou amoníaco[31] como fontes de poder redutor e obteñen enerxía da oxidación destes compostos utilizando como aceptores de electróns oxíxeno ou nitrito.[32] Estes procesos microbióticos son importantes nos ciclos bioxeoquímicos en procesos como a nitrificación e a desnitrificación, esenciais para a fertilidade do solo[33][34]
Enerxía da luz
[editar | editar a fonte]As plantas, cianobacterias , bacterias púrpuras, bacterias verdes do xofre e algas poden captar a enerxía da luz. Este proceso está ligado á conversión do dióxido de carbono en compostos orgánicos, como parte da fotosíntese. Porén, os sistemas de captura de enerxía e de fixación de carbono poden operar separadamente en procariotas, xa que as bacterias púrpuras e verdes do xofre poden usar a luz do sol como fonte de enerxía, mentres que poden cambiar entre a fixación do carbono autótrofa e a fermentación de compostos orgánicos.[35][36]
A captura de enerxía solar e a súa transformación en enerxía química é un proceso parecido á fosforilación oxidativa, xa que almacena enerxía en forma dun gradiente de protóns e intervén unha ATP sintase, que dá lugar á síntese de ATP.[12] Os electróns necesarios despréndense dos pigmentos fotosintéticos (clorofilas, bacterioclorofilas), que están agrupados nas proteínas formando fotosistemas. O electrón sae dunha parte do fotosistema chamado centro de reacción. Estas estruturas son clasificadas en dous tipos dependendo do seu pigmento fotosintético; as plantas e cianobacterias teñen os dous tipos e as outras bacterias só un.[37]
Nas plantas, o fotosistema II perde electróns ao darlle a luz que os recupera de moléculas de auga, proceso no que a molécula de auga rompe liberando oxíxeno como produto residual. Os electróns flúen despois cara ao complexo do citocromo b6f, que usa a súa enerxía para bombear protóns a través da membrana dos tilacoides do cloroplasto.[15] Estes protóns móvense a través da ATP-sintase do cloroplasto polo mesmo mecanismo explicado na fosforilación oxidativa. Os electróns logo flúen polo fotosistema I e poden ser utilizados para reducir o coencima NADP+ (que será utilizado no ciclo de Calvin) ou reciclados (fosforilación cíclica) para xerar máis ATP.[38]
Anabolismo
[editar | editar a fonte]- Artigo principal: Anabolismo.
O anabolismo é o conxunto de procesos metabólicos construtivos nos que se utiliza a enerxía liberada polo catabolismo para sintetizar moléculas complexas. En xeral, as moléculas complexas que dan lugar a estruturas celulares constrúense a partir de precursores simples. O anabolismo comprende tres facetas. Primeiro, a produción de precursores como os aminoácidos, monosacáridos, isoprenoides e nucleótidos; segundo, a súa activación como reactivos usando enerxía do ATP; e terceiro, a unión destes precursores en moléculas máis complexas como proteínas, polisacáridos, lípidos e ácidos nucleicos.
Os organismos difiren nos tipos de moléculas que poden sintetizar por si mesmos nas súas células. Os organismos autótrofos, como as plantas, poden construír moléculas orgánicas complexas e proteínas a partir de moléculas simples como dióxido de carbono, auga e sales minerais. Os organismos heterótrofos, polo contrario, precisan unha fonte de substancias orgánicas máis complexas, como monosacáridos e aminoácidos, para producir estas macromoléculas. Os organismos poden clasificarse pola fonte de enerxía utilizada en:
- Fotótrofos (fotoautótrofos e fotoheterótrofos), que obteñen a enerxía do Sol.
- Quimiótrofos (quimioheterótrofos e quimioautótrofos), que obteñen a energía mediante reaccións oxidativas de distintos compostos químicos.
Fixación do carbono
[editar | editar a fonte]
A fotosíntese é a síntese de glicosa a partir de enerxía solar, dióxido de carbono (CO2) e un doante de electróns, que nas plantas, algas e cianobacterias é a auga (H2O). Este proceso utiliza o ATP e o NADPH producidos pola fase luminosa fotosintética para usar o CO2 para formar 3-fosfoglicerato, que pode ser convertido en glicosa. Esta reacción de fixación do CO2 é levada a cabo polo encima RuBisCO como parte do ciclo de Calvin.[39] Nas plantas hai tres tipos posibles de fotosíntese segundo a forma de fixar o carbono, que son: C3, C4 e CAM. Segundo o tipo de que se trate a fixación do CO2 no ciclo de Calvin pode ser directa (desde o CO2 do aire), como ocorre na C3, ou indirecta (se este se fixa primeiro noutra molécula orgánica), como ocorre na C4 e CAM; estas dúas últimas son adaptacións para soportar a luz solar intensa e as condicións secas.[40]
En procariotas fotosintéticos os mecanismos da fixación son máis diversos. O CO2 pode ser fixado polo ciclo de Calvin, e polo ciclo de Krebs inverso,[41] ou a carboxilación do acetil-CoA.[42][43] Os quimioautótrofos tamén poden fixar o CO2 por medio do ciclo de Calvin, pero utilizan a enerxía de compostos inorgánicos para levar a cabo a reacción.[44]
- Véxase tamén: Fotosíntese, Fotorrespiración e Quimiosíntese.
Carbohidratos
[editar | editar a fonte]No anabolismo de carbohidratos, poden sintetizarse ácidos orgánicos simples a partir de monosacáridos como a glicosa e logo sintetizar polisacáridos como o amidón. A xeración de glicosa a partir de compostos como o piruvato, lactato, glicerol e aminoácidos denomínase gliconeoxénese. A gliconeoxénese transforma o piruvato en glicosa 6-fosfato a través dunha serie de intermediarios, moitos dos cales son os mesmos que os da glicólise.[22] Porén, esta ruta non é simplemente a inversa da glicólise, xa que varios pasos son catalizados por encimas non glicolíticos. Isto é importante á hora de evitar que ambas as rutas estean activas á vez dando lugar a un ciclo fútil.[45][46]
A pesar de que a graxa é unha forma común de almacenamento de enerxía, en xeral nos vertebrados como os humanos os ácidos graxos non poden ser transformados en glicosa por gliconeoxénese, xa que estes organismos non poden converter acetil-CoA en piruvato.[47] Como resultado, tras un tempo de inanición, os vertebrados vense obrigados a producir corpos cetónicos a partir de ácidos graxos para substituír a glicosa en tecidos como o cerebro, que non pode metabolizar ácidos graxos.[48] Noutros organismos como as plantas e as bacterias, este problema metabólico é solucionado utilizando o ciclo do glioxilato, que evita o paso de descarboxilación do ciclo de Krebs e permite a transformación de acetil-CoA en oxalacetato, o cal pode ser utilizado para a síntese de glicosa.[47][49]
Os polisacáridos son sintetizados por medio dunha adición secuencial de monosacáridos levada a cabo por glicosil-transferases desde un doante reactivo azucre-fosfato a un aceptor como o grupo hidroxilo no polisacárido que se sintetiza. Como calquera dos grupos hidroxilos do anel do composto pode ser aceptor, os polisacáridos producidos poden ter estruturas ramificadas ou liñais.[50] Estes polisacáridos producidos poden ter funcións metabólicas ou estruturais ou poden ser transferidos a lípidos e proteínas por medio de encimas.[51][52]
- Véxase tamén: Gliconeoxénese, Glicoxénese e Glicosilación.
Ácidos graxos, isoprenoides e esteroides
[editar | editar a fonte]- Artigos principais: Ácido graxo, Isoprenoide e Esteroide.

Os ácidos graxos sintetízanse ao polimerizar e reducir unidades de acetil-CoA. As cadeas nos ácidos graxos alóngadanse por medio dun ciclo de reaccións que engaden o grupo acetil, redúceno a alcohol, deshidratan un grupo alqueno e logo redúceno novamente a un grupo alcano. Os encimas que interveñen na síntese de ácidos graxos divídense en dous grupos: nos animais e fungos, as reaccións da síntese lévaas a cabo unha soa proteína multifuncional tipo I,[53] mentres que en plastidios de plantas e en bacterias son os encimas tipo II por separado os que levan a cabo cada paso da ruta.[54][55]
Os terpenos e isoprenoides son clases de lípidos entre os que están os carotenoides e forman a familia máis ampla de produtos naturais da planta.[56] Estes compostos son sintetizados pola unión e modificación de unidades de isopreno doadas polos precursores reactivos pirofosfosfatados isopentenil pirofosfato e dimetilalil pirofosfato.[57] Estes precursores poden sintetizarse de diversos modos. En animais e arqueas, estes compostos sintetízanse a partir de acetil-CoA,[58] mentres que en plantas e bacterias se fai a partir de piruvato e gliceraldehido 3-fosfato como substratos.[57][59] Unha reacción que usa estes doantes isoprénicos activados é a biosíntese de esteroides. neste caso, as unidades de isoprenoides únense covalentemente para formar escualeno, que se prega formando unha serie de aneis dando lugar a unha molécula denominada lanosterol.[60] O lanosterol pode logo ser transformado en esteroides como o colesterol.
Proteínas
[editar | editar a fonte]- Artigo principal: Síntese de proteínas.
A capacidade dos organismos de sintetizar os 20 aminoácidos proteinoxénicos é diversa. As bacterias e as plantas poden sintetizar os 20, pero os mamíferos poden sintetizar só 10.[8] Os outros denomínanse aminoácidos esenciais e deben obterse dos alimentos. Todos os aminoácidos son sintetizados a partir de intermediarios da glicólise e o ciclo de Krebs. O nitróxeno obtense do ácido glutámico e a glutamina. A síntese de aminoácidos depende da formación do alfa-cetoácido apropiado, que despois é transaminado para formar un aminoácido.[61]
Os aminoácidos dan lugar ás proteínas ao enlazarse por enlace peptídico e dar lugar a unha cadea. Cada proteína diferente ten unha secuencia única de aminoácidos (estrutura primaria). Os aminoácidos poden formar unha gran variedade de proteínas dependendo da secuencia destes na proteína. As proteínas están constituídas por aminoácidos que foron activados pola adición dun ARNt a través dun enlace éster.[62] O aminoacil-ARNt entra despois nun ribosoma, que vai engadindo os residuos de aminoácidos á cadea proteica, baseándose na secuencia de información que vai "lendo" nunha molécula de ARN mensaxeiro.[63]
Síntese de nucleótidos
[editar | editar a fonte]Os nucleótidos son sintetizados a partir de aminoácidos, dióxido de carbono e ácido fórmico en rutas que requiren unha cantidade grande de enerxía metabólica.[64][65] En consecuencia, a maioría dos organismos teñen un sistema eficiente para conservar os nucleótidos preformados.[64][66] As purinas sintetízanse como nucleósidos (bases unidas a ribosa). Tanto a adenina coma a guanina sintetízanse a partir dun precursor nucleósido, a inosina monofosfato, que se sintetiza usando átomos dos aminoácidos glicina, glutamina e ácido aspártico; tamén ocorre o mesmo co HCOO−, que é transferido desde o coencima tetrahidrofolato. As pirimidinas, polo contrario, sintetízanse a partir de ácido orótico, que á súa vez se sintetiza a partir da glutamina e o aspartato.[67]
- Véxase tamén: Replicación do ADN e Transcrición xenética.
Biosíntese de metabolitos secundarios
[editar | editar a fonte]- Artigo principal: Metabolito secundario.
A serie de procesos metabólicos implicados nas funcións vitais dos organismos denomínase metabolismo primario. Existe tamén un conxunto de reaccións bioquímicas denominado metabolismo secundario, que se producen de forma paralela ao metabolismo primario. Os compostos orgánicos producidos (metabolitos secundarios) non teñen un papel directo no crecemento ou reprodución dos seres vivos senón que cumpren funcións complementarias das vitais, tales como a comunicación intra e interespecífica (como no caso dos pigmentos aposemáticos e os aleloquímicos), protección contra condicións de estrés ambiental (tales como radiación, conxelación, seca e estrés salino) e ataque de depredadores, patóxenos ou parasitos (como no caso das fitotoxinas, antibióticos e fitoalexinas). As principais rutas metabólicas secundarias son as rutas do mevalonato e da 5-fosfono-1-desoxi-D-xilulosa, a ruta do acetato-malonato, a ruta do ácido shiquímico e as rutas secundarias de aminoácidos.[68].
Xenobióticos e metabolismo redutor
[editar | editar a fonte]- Artigo principal: Xenobiótico.
Todos os organismos están constanmente expostos a compostos e elementos químicos que non poden utilizar como alimento e serían daniños se se acumulasen nas súas células, xa que non terían unha función metabólica. Estes compostos potencialmente nocivos chámanse xenobióticos.[69] Os xenobióticos como as drogas sintéticas, os velenos naturais e os antibióticos son detoxificados por un conxunto de encimas metabolizadores de xenobióticos. Nos humanos, entre eles están as citocromo P450 oxidases,[70] as UDP-glicuroniltransferases[71] e as glutatión-S-transferases.[72]
Este sistema de encimas actúa en tres fases. En primeiro lugar, oxida os xenobióticos (fase I) e logo conxuga na molécula grupos solubles en auga (fase II). O xenobiótico modificado pode extraerse da célula por exocitose e, en organismos pluricelulares, pode ser metabolizado máis antes de ser excretado (fase III). En ecoloxía, estas reaccións son particularmente importantes pola biodegradación microbiana de axentes contaminantes e para a biorremediación de terras contaminadas.[73] Moitas destas reaccións microbióticas son compartidas con organismos pluricelulares, pero debido á maior biodiversidade dos microbios, estes poden tratar unha gama máis ampla de xenobióticos en comparación cos organismos pluricelulares; os microbios poden mesmo degradar axentes contaminantes como os compostos organoclorados.[74]
Un problema relacionado cos organismos aeróbicos é o estrés oxidativo.[75] Porén, unha bacteria estresada podería ser máis efectiva para a degradación destes contaminantes.[76]
Os procesos como a fosforilación oxidativa e a formación de enlaces disulfuro durante o pregamento das proteínas producen especies reactivas do oxíxeno como o peróxido de hidróxeno.[77] Estes oxidantes daniños son neutralizados por metabolitos antioxidantes como o glutatión e por encimas como as catalases e as peroxidases.[78][79]
Un exemplo de metabolismo xenobiótico é a depuración dos fármacos por parte do fígado.
Homeostase: regulación e control
[editar | editar a fonte]- Artigo principal: Homeostase.
Debido a que o ambiente dos organismos cambia constantemente, as reaccións metabólicas deben ser reguladas para manter unhas determinadas condicións na célula, o que se denomina homeostase.[80][81] Esta regulación permite aos organismos responder a estímulos e interaccionar co ambiente.[82] Para entendermos como se controlan as vías metabólicas, existen dous conceptos vencellados. En primeiro lugar, a regulación dun encima nunha ruta consiste en incrementar ou diminuír a súa actividade en resposta a sinais ou estímulos. En segundo lugar, o control levado a cabo por este encima depende dos efectos que teñen ditos cambios na súa actividade sobre a velocidade da ruta (o fluxo da ruta).[83] Por exemplo, un encima mostra cambios na súa actividade; pero se estes cambios teñen un efecto mínimo no fluxo da ruta metabólica, entón este encima non intervén no control da ruta.[84]
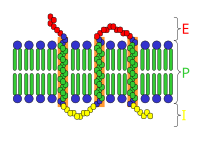
E: espazo extracelular.
P: membrana plasmática.
I: espazo intracelular.
Existen múltiples niveis nos que se pode regular o metabolismo. Na regulación intrínseca, a ruta metabólica autorregúlase para responder a cambios nos niveis de substratos ou produtos; por exemplo, unha diminución na cantidade de produtos pode incrementar o fluxo na ruta para compensalo.[83] Este tipo de regulación adoita implicar unha regulación alostérica das actividades dos distintos encimas da ruta.[85] O control extrínseco implica a unha célula nun organismo pluricelular, que cambia o seu metabolismo en resposta a sinais doutras células. Estes sinais son enviados xeralmente en forma de mensaxeiros como as hormonas e os factores de crecemento, que son detectados por receptores celulares específicos na superficie da célula.[86] Estes sinais son transmitidos cara ao interior da célula mediante mensaxeiros secundarios que xeralmente implican a fosforilación de proteínas.[87]
Un exemplo de control extrínseco é a regulación do metabolismo da glicosa por medio da hormona denominada insulina.[88] A insulina prodúcese como consecuencia dun aumento da concentración de azucre (glicosa) no sangue. A unión desta hormona aos receptores de insulina activa unha fervenza de proteína-quinases que estimulan a absorción de glicosa por parte da célula para transformala en moléculas de almacenamento como os ácidos graxos e o glicóxeno.[89] O metabolismo do glicóxeno é controlado pola actividade da glicóxeno fosforilase, encima que degrada o glicóxeno, e a glicóxeno sintase, encima que o sintetiza. Estes encimas son regulados dun modo recíproco, de modo que a fosforilación inhibe a glicóxeno sintase, pero activa á súa vez a glicóxeno fosforilase. A insulina induce a síntese de glicóxeno ao activar fosfatases e producir unha diminución na fosforilación destes encimas.[90]
- Véxase tamén: Hormona e Comunicación celular.
Termodinámica dos organismos vivos
[editar | editar a fonte]Os organismos vivos deben respectar as leis da termodinámica. A segunda lei da termodinámica establece que en calquera sistema pechado, a cantidade de entropía terá unha tendencia a aumentar. A pesar de que a complexidade dos organismos vivos contradice aparentemente esta lei, a vida é posible porque todos os organismos vivos son sistemas abertos que intercambian materia e enerxía co medio que os rodea. Os sistemas vivos non están en equilibrio, senón que son sistemas de disipación que manteñen o seu estado de complexidad ao provocaren incrementos maiores na entropía do medioque os rodea.[91] O metabolismo dunha célula consegue isto por medio da relación entre os procesos espontáneos do catabolismo cos procesos non espontáneos do anabolismo. En termos termodinámicos, o metabolismo mantén a orde ao crear desorde.[92]
- Véxase tamén: Enerxía libre de Gibbs.
Investigación e manipulación
[editar | editar a fonte]
Clasicamente, o metabolismo estúdase facendo unha aproximación reducionista centrada nunha ruta metabólica específica. É especialmente útil o uso de trazadores radioactivos no organismo completo, ou en tecidos ou células, para determinar as rutas desde os precursores aos produtos finais identificando os intermediarios marcados radiactivamente e os produtos.[93] Os encimas que catabolizan estas reaccións químicas poden purificarse para estudar a súa cinética encimática e as respostas que presentan ante diversos inhibidores. Outro tipo de estudo que se pode levar a cabo en paralelo é a identificación dos metabolitos presentes nunha célula ou tecido; o estudo de todo o conxunto destas moléculas denomínase metabolómica. Estes estudos ofrecen unha visión das estruturas e funcións de rutas metabólicas simples, mais son pouco axeitados para aplicalos a sistemas máis complexos como o metabolismo global da célula.[94]
Na imaxe da dereita pódese apreciar a complexidade dunha rede metabólica celular que mostra interaccións entre só 43 proteínas e 40 metabolitos (nunha célula real serían miles). As secuencias de xenomas de plantas proporcionan listas que conteñen ata 45.000 xenes.[95] Porén, é posible usar esta información para reconstruír redes completas de reaccións bioquímicas e producir máis modelos matemáticos holísticos que poidan explicar e predicir o seu comportamento.[96] Estes modelos son moito máis efectivos cando se usan para integrar a información obtida das rutas e dos metabolitos polos métodos clásicos cos datos de expresión xénica obtidos por medio de estudos de proteómica e de chips de ADN.[97]
Unha das aplicacións tecnolóxicas desta información é a enxeñaría metabólica. Con esta tecnoloxía, pódense modificar xeneticamente organismos como os lévedos, as plantas ou as bacterias para facelos máis útiles nalgún eido da biotecnoloxía, como pode ser a produción de fármacos, antibióticos ou substancias químicas industriais.[98][99][100] Estas modificacións xenéticas teñen como obxectivo reducir a cantidade de enerxía usada para producir o produto, incrementar os beneficios e reducir a produción de refugallos.[101]
- Véxase tamén: Metabolómica, Proteómica, Cinética encimática e Inhibidor encimático.
Evolución
[editar | editar a fonte]- Artigos principais: Evolución , Evolución molecular e Filoxenética.

As rutas centrais do catabolismo descritas antes, como a glicólise e o ciclo de Krebs, están presentes nos tres dominios de seres vivos e seguramente estaban xa presentes no antepasado universal común.[3][103] Esta célula antepasada universal dos seres vivos era procariota e probablemente un organismo metanóxeno que tiña un amplo metabolismo de aminoácidos, nucleótidos, carbohidratos e lípidos.[104][105] A conservación destas vías metabólicas primitivas durante as posteriores fase da evoloución pode ser o resultado de que estas reaccións son unha óptima solución aos seus problemas metabólicos particulares, e vías como a glicólise e o ciclo do ácido cítrico producen os seus produtos finais con alta eficiencia e nun número mínimo de pasos.[4][5][106] As primeiras vías metabólicas puideron ser parte do antigo mundo de ARN, e as primeiras vías do metabolismo baseado en encimas puideron ser parte do metabolismo das purinas.[107]
Propuxéronse moitos modelos para describir os mecanismos polos cales evolucionaron as novas vías metabólicas. Entre eles está a adición secuencial de novos encimas a unha curta vía ancestral, a duplicación e despois a diverxencia de vías enteiras e o recrutamento de encimas preexistentes e a súa ensamblaxe en vías con novas reaccións.[108] A importancia relativa destes mecanismos non está claro, pero os estudos xenómicos mostran que os encimas dunha vía evolucionaron probablemente a partir dun antepasado compartido, e suxiren que moitas vías evolucionaron paso a paso e fóronse creando novas funcións a a partir de pasos preexistentes na vía.[109] Un modelo alternativo procede dos estudos que tratan de rastrear a evolución das estruturas das proteínas nas redes metabólicas, e propón que os encimas son recrutados de forma xeneralizada, tomando prestados encimas de vías metabólicas diferentes para realizar funcións similares noutras vías (evidente na base de datos MANET)[110] Este proceso de recrutamento orixinou un mosaico encimático evolutivo.[111] Unha terceira posibilidade é que algunhas partes do metabolismo poderían existir como "módulos" que poden reutilizarse en diferentes rutas e realizar similares funcións sobre distintas moléculas.[112]
Igual que a evolución orixina novas rutas metabólicas, esta pode tamén causar a perda de funcións metabólicas. Por exemplo, perdéronse algúns procesos metabólicos de parasitos que non son esenciais para sobrevivir e como alternativa poden captar do seu hóspede aminoácidos, nucleótidos e carbohidratos xa formados.[113] Poden verse tamén capacidades metabólicas reducidas en organismos endosimbiontes.[114]
Historia
[editar | editar a fonte]
O termo metabolismo deriva do grego Μεταβολισμός – "Metabolismos", "cambio", ou "derrocamento".[115] A historia do estudo científico do metabolismo abrangue moitos séculos e empezou examinando animais completos, e seguiu en tempos da moderna bioquímica examinando as reaccións metabólicas por separado. Os primeiros experimentos controlados sobre metabolismo humano publicounos Santorio Santorio en 1614 no seu libro Ars de statica medicina.[116] Alí describía como se pesou antes e despois de comer, durmir, traballar, ter relacións sexuais, xaxuar, beber, e excretar. A conclusión foi que a maioría da comida que tomaba se perdía no que el chamaba "transpiración insensible".
Nestes primeiros estudos, non se identificaron os mecanismos destes procesos metabólicos e pensábase que había unha forza vital que animaba os tecidos vivos.[117] No século XIX, cando se estudou a fermentación do azucre a alcohol realizado polos lévedos, Louis Pasteur tirou a conclusión de que dita fermentación estaba catalizada por substancias contidas nos lévedos, que el chamou "fermentos". Escribiu: "a fermentación alcohólica é un acto correlacionado coa vida e a organización das células de lévedos, non coa morte e a putrefacción das células."[118] Este descubrimento, xunto coa publicación por Friedrich Wöhler en 1828 da síntese química da urea,[119] que foi o primeiro composto orgánico obtido utilizando precursores inorgánicos, probou que os compostos orgánicos e as reaccións químicas que se dan nas células non eran diferentes, en principio, ás de calquera outra rama da química.
O descubrimento dos encimas por Eduard Buchner a principios do século XX permitiu separar o estudo das reaccións químicas metabólicas do estudo biolóxico das células, e significou o comezo da bioquímica.[120] Os coñecementos bioquímicos medraron rapidamente ao longo do século XX. Un dos máis prolíficos dos modernos bioquímicos foi Hans Krebs que fixo grandes contribucións ao estudo do metabolismo.[121] Krebs descubriu o ciclo da urea e despois, traballando con Hans Kornberg, o ciclo do ácido cítrico e o do glioxilato.[122][49] A moderna investigación bioquímica viuse moi pulada polo desenvolvemento de novas técnicas como a cromatografía, difracción de raios X, espectroscopía de resonancia magnética nuclear, marcaxe radioisotópica, microscopía electrónica e simulacións de dinámica molecular. Estas técnicas permitiron o descubrimento e análise detallada de moitas moléculas e vías metabólicas das células.
Notas
[editar | editar a fonte]- ↑ 1,0 1,1 Friedrich C (1998). "Physiology and genetics of sulfur-oxidizing bacteria". Adv Microb Physiol. Advances in Microbial Physiology 39: 235–89. ISBN 9780120277391. PMID 9328649. doi:10.1016/S0065-2911(08)60018-1.
- ↑ Pace NR (2001). "The universal nature of biochemistry". Proc. Natl. Acad. Sci. U.S.A. 98 (3): 805–8. Bibcode:2001PNAS...98..805P. PMC 33372. PMID 11158550. doi:10.1073/pnas.98.3.805.
- ↑ 3,0 3,1 Smith E, Morowitz H (2004). "Universality in intermediary metabolism". Proc Natl Acad Sci USA 101 (36): 13168–73. Bibcode:2004PNAS..10113168S. PMC 516543. PMID 15340153. doi:10.1073/pnas.0404922101. Arquivado dende o orixinal o 02 de xuño de 2020. Consultado o 16 de xullo de 2013.
- ↑ 4,0 4,1 Ebenhöh O, Heinrich R (2001). "Evolutionary optimization of metabolic pathways. Theoretical reconstruction of the stoichiometry of ATP and NADH producing systems". Bull Math Biol 63 (1): 21–55. PMID 11146883. doi:10.1006/bulm.2000.0197.
- ↑ 5,0 5,1 Meléndez-Hevia E, Waddell T, Cascante M (1996). "The puzzle of the Krebs citric acid cycle: assembling the pieces of chemically feasible reactions, and opportunism in the design of metabolic pathways during evolution". J Mol Evol 43 (3): 293–303. PMID 8703096. doi:10.1007/BF02338838.
- ↑ Todar's Online Textbook of bacteriology. Diversity of Metabolism in Procaryotes (page 5) [1]
- ↑ Gobierno de Canarias (ed.). "La célula, estructura y fisiología" (PDF). Consejería de Educación. Arquivado dende o orixinal (PDF) o 02-12-2006. Consultado o 26-10-2007.
- ↑ 8,0 8,1 Nelson DL, Cox MM (2005). Lehninger Principles of Biochemistry. New York: W. H. Freeman and company. p. 841. ISBN 0-7167-4339-6.
- ↑ 9,0 9,1 Wimmer M, Rose I (1978). "Mechanisms of enzyme-catalyzed group transfer reactions". Annu Rev Biochem 47: 1031–78. PMID 354490.
- ↑ Heymsfield S, Waki M, Kehayias J, Lichtman S, Dilmanian F, Kamen Y, Wang J, Pierson R (1991). "Chemical and elemental analysis of humans in vivo using improved body composition models". Am J Physiol 261 (2 Pt 1): E190–8. PMID 1872381.
- ↑ Mitchell P (1979). "The Ninth Sir Hans Krebs Lecture. Compartmentation and communication in living systems. Ligand conduction: a general catalytic principle in chemical, osmotic and chemiosmotic reaction systems". Eur J Biochem 95 (1): 1–20. PMID 378655.
- ↑ 12,0 12,1 12,2 12,3 Dimroth P, von Ballmoos C, Meier T (2006). "Catalytic and mechanical cycles in F-ATP synthases. Fourth in the Cycles Review Series". EMBO Rep 7 (3): 276–82. PMID 16607397.[Ligazón morta]
- ↑ Pollak N, Dölle C, Ziegler M (2007). "The power to reduce: pyridine nucleotides--small molecules with a multitude of functions". Biochem J 402 (2): 205–18. PMID 17295611.
- ↑ Nealson K, Conrad P (1999). "Life: past, present and future". Philos Trans R Soc Lond B Biol Sci 354 (1392): 1923–39. PMID 10670014.[Ligazón morta]
- ↑ 15,0 15,1 Nelson N, Ben-Shem A (2004). "The complex architecture of oxygenic photosynthesis". Nat Rev Mol Cell Biol 5 (12): 971–82. PMID 15573135.
- ↑ Häse C, Finkelstein R (1993). "Bacterial extracellular zinc-containing metalloproteases". Microbiol Rev 57 (4): 823–37. PMID 8302217.[Ligazón morta]
- ↑ Gupta R, Gupta N, Rathi P (2004). "Bacterial lipases: an overview of production, purification and biochemical properties". Appl Microbiol Biotechnol 64 (6): 763–81. PMID 14966663.
- ↑ Hoyle T (1997). "The digestive system: linking theory and practice". Br J Nurs 6 (22): 1285–91. PMID 9470654.
- ↑ Souba W, Pacitti A (1992). "How amino acids get into cells: mechanisms, models, menus, and mediators". JPEN J Parenter Enteral Nutr 16 (6): 569–78. PMID 1494216.
- ↑ Barrett M, Walmsley A, Gould G (1999). "Structure and function of facilitative sugar transporters". Curr Opin Cell Biol 11 (4): 496–502. PMID 10449337.
- ↑ Bell G, Burant C, Takeda J, Gould G (1993). "Structure and function of mammalian facilitative sugar transporters". J Biol Chem 268 (26): 19161–4. PMID 8366068.
- ↑ 22,0 22,1 Bouché C, Serdy S, Kahn C, Goldfine A (2004). "The cellular fate of glucose and its relevance in type 2 diabetes". Endocr Rev 25 (5): 807–30. PMID 15466941. Arquivado dende o orixinal o 04 de decembro de 2012. Consultado o 12 de xullo de 2013.
- ↑ Sakami W, Harrington H (1963). "Amino acid metabolism". Annu Rev Biochem 32: 355–98. PMID 14144484.
- ↑ Brosnan J (2000). "Glutamate, at the interface between amino acid and carbohydrate metabolism". J Nutr 130 (4S Suppl): 988S–90S. PMID 10736367.
- ↑ Young V, Ajami A (2001). "Glutamine: the emperor or his clothes?". J Nutr 131 (9 Suppl): 2449S–59S; discussion 2486S–7S. PMID 11533293.
- ↑ Hosler J, Ferguson-Miller S, Mills D (2006). "Energy transduction: proton transfer through the respiratory complexes". Annu Rev Biochem 75: 165–87. PMID 16756489.
- ↑ Schultz B, Chan S (2001). "Structures and proton-pumping strategies of mitochondrial respiratory enzymes". Annu Rev Biophys Biomol Struct 30: 23–65. PMID 11340051.
- ↑ Capaldi R, Aggeler R (2002). "Mechanism of the F(1)F(0)-type ATP synthase, a biological rotary motor". Trends Biochem Sci 27 (3): 154–60. PMID 11893513.
- ↑ Friedrich B, Schwartz E (1993). "Molecular biology of hydrogen utilization in aerobic chemolithotrophs". Annu Rev Microbiol 47: 351–83. PMID 8257102.
- ↑ Weber K, Achenbach L, Coates J (2006). "Microorganisms pumping iron: anaerobic microbial iron oxidation and reduction". Nat Rev Microbiol 4 (10): 752–64. PMID 16980937.
- ↑ Jetten M, Strous M, van de Pas-Schoonen K, Schalk J, van Dongen U, van de Graaf A, Logemann S, Muyzer G, van Loosdrecht M, Kuenen J (1998). "The anaerobic oxidation of ammonium". FEMS Microbiol Rev 22 (5): 421–37. PMID 9990725.
- ↑ Simon J (2002). "Enzymology and bioenergetics of respiratory nitrite ammonification". FEMS Microbiol Rev 26 (3): 285–309. PMID 12165429.
- ↑ Conrad R (1996). "Soil microorganisms as controllers of atmospheric trace gases (H2, CO, CH4, OCS, N2O, and NO)". Microbiol Rev 60 (4): 609–40. PMID 8987358.[Ligazón morta]
- ↑ Barea J, Pozo M, Azcón R, Azcón-Aguilar C (2005). "Microbial co-operation in the rhizosphere". J Exp Bot 56 (417): 1761–78. PMID 15911555. Arquivado dende o orixinal o 16 de febreiro de 2007. Consultado o 12 de xullo de 2013.
- ↑ van der Meer M, Schouten S, Bateson M, Nübel U, Wieland A, Kühl M, de Leeuw J, Sinninghe Damsté J, Ward D (2005). "Diel variations in carbon metabolism by green nonsulfur-like bacteria in alkaline siliceous hot spring microbial mats from Yellowstone National Park". Appl Environ Microbiol 71 (7): 3978–86. PMID 16000812.[Ligazón morta]
- ↑ Tichi M, Tabita F (2001). "Interactive control of Rhodobacter capsulatus redox-balancing systems during phototrophic metabolism". J Bacteriol 183 (21): 6344–54. PMID 11591679.[Ligazón morta]
- ↑ Allen J, Williams J (1998). "Photosynthetic reaction centers". FEBS Lett 438 (1-2): 5–9. PMID 9821949.
- ↑ Munekage Y, Hashimoto M, Miyake C, Tomizawa K, Endo T, Tasaka M, Shikanai T (2004). "Cyclic electron flow around photosystem I is essential for photosynthesis". Nature 429 (6991): 579–82. PMID 15175756.
- ↑ Miziorko H, Lorimer G (1983). "Ribulose-1,5-bisphosphate carboxylase-oxygenase". Annu Rev Biochem 52: 507–35. PMID 6351728.
- ↑ Dodd A, Borland A, Haslam R, Griffiths H, Maxwell K (2002). "Crassulacean acid metabolism: plastic, fantastic". J Exp Bot 53 (369): 569–80. PMID 11886877. Arquivado dende o orixinal o 25 de xaneiro de 2009. Consultado o 12 de xullo de 2013.
- ↑ Hügler M, Wirsen C, Fuchs G, Taylor C, Sievert S (2005). "Evidence for autotrophic CO2 fixation via the reductive tricarboxylic acid cycle by members of the epsilon subdivision of proteobacteria". J Bacteriol 187 (9): 3020–7. PMID 15838028.[Ligazón morta]
- ↑ Strauss G, Fuchs G (1993). "Enzymes of a novel autotrophic CO2 fixation pathway in the phototrophic bacterium Chloroflexus aurantiacus, the 3-hydroxypropionate cycle". Eur J Biochem 215 (3): 633–43. PMID 8354269.
- ↑ Wood H (1991). "Life with CO or CO2 and H2 as a source of carbon and energy". FASEB J 5 (2): 156–63. PMID 1900793.
- ↑ Shively J, van Keulen G, Meijer W (1998). "Something from almost nothing: carbon dioxide fixation in chemoautotrophs". Annu Rev Microbiol 52: 191–230. PMID 9891798.
- ↑ Boiteux A, Hess B (1981). "Design of glycolysis". Philos Trans R Soc Lond B Biol Sci 293 (1063): 5–22. PMID 6115423.
- ↑ Pilkis S, el-Maghrabi M, Claus T (1990). "Fructose-2,6-bisphosphate in control of hepatic gluconeogenesis. From metabolites to molecular genetics". Diabetes Care 13 (6): 582–99. PMID 2162755.
- ↑ 47,0 47,1 Ensign S (2006). "Revisiting the glyoxylate cycle: alternate pathways for microbial acetate assimilation". Mol Microbiol 61 (2): 274–6. PMID 16856935.
- ↑ Finn P, Dice J. "Proteolytic and lipolytic responses to starvation". Nutrition 22 (7-8): 830–44. PMID 16815497.
- ↑ 49,0 49,1 Kornberg H, Krebs H (1957). "Synthesis of cell constituents from C2-units by a modified tricarboxylic acid cycle". Nature 179 (4568): 988–91. PMID 13430766.
- ↑ Rademacher T, Parekh R, Dwek R (1988). "Glycobiology". Annu Rev Biochem 57: 785–838. PMID 3052290.
- ↑ Opdenakker G, Rudd P, Ponting C, Dwek R (1993). "Concepts and principles of glycobiology". FASEB J 7 (14): 1330–7. PMID 8224606.
- ↑ McConville M, Menon A (2000). "Recent developments in the cell biology and biochemistry of glycosylphosphatidylinositol lipids (review)". Mol Membr Biol 17 (1): 1–16. PMID 10824734.
- ↑ Chirala S, Wakil S (2004). "Structure and function of animal fatty acid synthase". Lipids 39 (11): 1045–53. PMID 15726818.
- ↑ White S, Zheng J, Zhang Y (2005). "The structural biology of type II fatty acid biosynthesis". Annu Rev Biochem 74: 791–831. PMID 15952903.
- ↑ Ohlrogge J, Jaworski J (1997). "Regulation of fatty acid synthesis". Annu Rev Plant Physiol Plant Mol Biol 48: 109–136. PMID 15012259.
- ↑ Dubey V, Bhalla R, Luthra R (2003). "An overview of the non-mevalonate pathway for terpenoid biosynthesis in plants" (PDF). J Biosci 28 (5): 637–46. PMID 14517367.
- ↑ 57,0 57,1 Kuzuyama T, Seto H (2003). "Diversity of the biosynthesis of the isoprene units". Nat Prod Rep 20 (2): 171–83. PMID 12735695.
- ↑ Grochowski L, Xu H, White R (2006). "Methanocaldococcus jannaschii uses a modified mevalonate pathway for biosynthesis of isopentenyl diphosphate". J Bacteriol 188 (9): 3192–8. PMID 16621811.[Ligazón morta]
- ↑ Lichtenthaler H (1999). "The 1-Ddeoxy-D-xylulose-5-phosphate pathway of isoprenoid biosynthesis in plants". Annu Rev Plant Physiol Plant Mol Biol 50: 47–65. PMID 15012203.
- ↑ Schroepfer G (1981). "Sterol biosynthesis". Annu Rev Biochem 50: 585–621. PMID 7023367.
- ↑ Guyton, Arthur C.; John E. Hall (2006). Elsevier, ed. Textbook of Medical Physiology. Philadelphia. pp. 855-6. ISBN 0-7216-0240-1.
- ↑ Ibba M, Söll D (2001). "The renaissance of aminoacyl-tRNA synthesis". EMBO Rep 2 (5): 382-7. PMID 11375928. Arquivado dende o orixinal o 01 de maio de 2011. Consultado o 26 de agosto de 2018.
- ↑ Lengyel P, Söll D (1969). "Mechanism of protein biosynthesis". Bacteriol Rev 33 (2): 264–301. PMID 4896351.[Ligazón morta]
- ↑ 64,0 64,1 Rudolph F (1994). "The biochemistry and physiology of nucleotides". J Nutr 124 (1 Suppl): 124S–127S. PMID 8283301.
- ↑ Zrenner R, Stitt M, Sonnewald U, Boldt R (2006). "Pyrimidine and purine biosynthesis and degradation in plants". Annu Rev Plant Biol 57: 805–36. PMID 16669783.
- ↑ Stasolla C, Katahira R, Thorpe T, Ashihara H (2003). "Purine and pyrimidine nucleotide metabolism in higher plants". J Plant Physiol 160 (11): 1271–95. PMID 14658380.
- ↑ Smith J (1995). "Enzymes of nucleotide synthesis". Curr Opin Struct Biol 5 (6): 752–7. PMID 8749362.
- ↑ Paul M. Dewick (2009). John Wiley and Sons, ed. Medicinal natural products: a biosynthetic approach. ISBN 9780470741689.
- ↑ Testa B, Krämer S (2006). "The biochemistry of drug metabolism--an introduction: part 1. Principles and overview". Chem Biodivers 3 (10): 1053–101. PMID 17193224.
- ↑ Danielson P (2002). "The cytochrome P450 superfamily: biochemistry, evolution and drug metabolism in humans". Curr Drug Metab 3 (6): 561–97. PMID 12369887.
- ↑ King C, Rios G, Green M, Tephly T (2000). "UDP-glucuronosyltransferases". Curr Drug Metab 1 (2): 143–61. PMID 11465080.
- ↑ Sheehan D, Meade G, Foley V, Dowd C (2001). "Structure, function and evolution of glutathione transferases: implications for classification of non-mammalian members of an ancient enzyme superfamily". Biochem J 360 (Pt 1): 1–16. PMID 11695986.[Ligazón morta]
- ↑ Galvão T, Mohn W, de Lorenzo V (2005). "Exploring the microbial biodegradation and biotransformation gene pool". Trends Biotechnol 23 (10): 497–506. PMID 16125262.
- ↑ Janssen D, Dinkla I, Poelarends G, Terpstra P (2005). "Bacterial degradation of xenobiotic compounds: evolution and distribution of novel enzyme activities". Environ Microbiol 7 (12): 1868–82. PMID 16309386.
- ↑ Davies K (1995). "Oxidative stress: the paradox of aerobic life". Biochem Soc Symp 61: 1–31. PMID 8660387.
- ↑ Una bacteria "estresada" puede ser más eficiente Arquivado 09 de abril de 2008 en Wayback Machine. - La Nación, 24 de mayo de 2006.
- ↑ Tu B, Weissman J (2004). "Oxidative protein folding in eukaryotes: mechanisms and consequences". J Cell Biol 164 (3): 341–6. PMID 14757749.
- ↑ Sies H (1997). "Oxidative stress: oxidants and antioxidants" (PDF). Exp Physiol 82 (2): 291–5. PMID 9129943. Arquivado dende o orixinal (PDF) o 25 de marzo de 2009. Consultado o 12 de xullo de 2013.
- ↑ Vertuani S, Angusti A, Manfredini S (2004). "The antioxidants and pro-antioxidants network: an overview". Curr Pharm Des 10 (14): 1677–94. PMID 15134565.
- ↑ Albert R (2005). "Scale-free networks in cell biology". J Cell Sci 118 (Pt 21): 4947–57. PMID 16254242.
- ↑ Brand M (1997). "Regulation analysis of energy metabolism". J Exp Biol 200 (Pt 2): 193–202. PMID 9050227.
- ↑ Soyer O, Salathé M, Bonhoeffer S (2006). "Signal transduction networks: topology, response and biochemical processes". J Theor Biol 238 (2): 416–25. PMID 16045939.
- ↑ 83,0 83,1 Salter M, Knowles R, Pogson C (1994). "Metabolic control". Essays Biochem 28: 1–12. PMID 7925313.
- ↑ Westerhoff H, Groen A, Wanders R (1984). "Modern theories of metabolic control and their applications (review)". Biosci Rep 4 (1): 1–22. PMID 6365197.
- ↑ Fell D, Thomas S (1995). "Physiological control of metabolic flux: the requirement for multisite modulation". Biochem J. 311 (Pt 1): 35–9. PMID 7575476.[Ligazón morta]
- ↑ Hendrickson W (2005). "Transduction of biochemical signals across cell membranes". Q Rev Biophys 38 (4): 321–30. PMID 16600054.
- ↑ Cohen P (2000). "The regulation of protein function by multisite phosphorylation--a 25 year update". Trends Biochem Sci 25 (12): 596–601. PMID 11116185.
- ↑ Lienhard G, Slot J, James D, Mueckler M (1992). "How cells absorb glucose". Sci Am 266 (1): 86–91. PMID 1734513.
- ↑ Roach P (2002). "Glycogen and its metabolism". Curr Mol Med 2 (2): 101–20. PMID 11949930.
- ↑ Newgard C, Brady M, O'Doherty R, Saltiel A (2000). "Organizing glucose disposal: emerging roles of the glycogen targeting subunits of protein phosphatase-1" (PDF). Diabetes 49 (12): 1967–77. PMID 11117996.
- ↑ von Stockar U, Liu J (1999). "Does microbial life always feed on negative entropy? Thermodynamic analysis of microbial growth". Biochim Biophys Acta 1412 (3): 191–211. PMID 10482783.
- ↑ Demirel Y, Sandler S (2002). "Thermodynamics and bioenergetics". Biophys Chem 97 (2-3): 87–111. PMID 12050002.
- ↑ Rennie M (1999). "An introduction to the use of tracers in nutrition and metabolism". Proc Nutr Soc 58 (4): 935–44. PMID 10817161.
- ↑ Phair R (1997). "Development of kinetic models in the nonlinear world of molecular cell biology". Metabolism 46 (12): 1489–95. PMID 9439549.
- ↑ Sterck L, Rombauts S, Vandepoele K, Rouzé P, Van de Peer Y (2007). "How many genes are there in plants (... and why are they there)?". Curr Opin Plant Biol 10 (2): 199–203. PMID 17289424.
- ↑ Borodina I, Nielsen J (2005). "From genomes to in silico cells via metabolic networks". Curr Opin Biotechnol 16 (3): 350–5. PMID 15961036.
- ↑ Gianchandani E, Brautigan D, Papin J (2006). "Systems analyses characterize integrated functions of biochemical networks". Trends Biochem Sci 31 (5): 284–91. PMID 16616498.
- ↑ Thykaer J, Nielsen J (2003). "Metabolic engineering of beta-lactam production". Metab Eng 5 (1): 56–69. PMID 12749845..
- ↑ González-Pajuelo M, Meynial-Salles I, Mendes F, Andrade J, Vasconcelos I, Soucaille P. "Metabolic engineering of Clostridium acetobutylicum for the industrial production of 1,3-propanediol from glycerol". Metab Eng 7 (5-6): 329–36. PMID 16095939.
- ↑ Krämer M, Bongaerts J, Bovenberg R, Kremer S, Müller U, Orf S, Wubbolts M, Raeven L (2003). "Metabolic engineering for microbial production of shikimic acid". Metab Eng 5 (4): 277–83. PMID 14642355.
- ↑ Koffas M, Roberge C, Lee K, Stephanopoulos G (1999). "Metabolic engineering". Annu Rev Biomed Eng 1: 535–57. PMID 11701499.
- ↑ Ciccarelli FD, Doerks T, von Mering C, Creevey CJ, Snel B, Bork P (2006). "Toward automatic reconstruction of a highly resolved tree of life". Science 311 (5765): 1283–7. PMID 16513982. doi:10.1126/science.1123061.
- ↑ Romano A, Conway T (1996). "Evolution of carbohydrate metabolic pathways". Res Microbiol 147 (6–7): 448–55. PMID 9084754. doi:10.1016/0923-2508(96)83998-2.
- ↑ Koch A (1998). "How did bacteria come to be?". Adv Microb Physiol. Advances in Microbial Physiology 40: 353–99. ISBN 978-0-12-027740-7. PMID 9889982. doi:10.1016/S0065-2911(08)60135-6.
- ↑ Ouzounis C, Kyrpides N (1996). "The emergence of major cellular processes in evolution". FEBS Lett 390 (2): 119–23. PMID 8706840. doi:10.1016/0014-5793(96)00631-X.
- ↑ C.Michael Hogan. 2010. Mutation. ed. E.Monosson and C.J.Cleveland. Encyclopedia of Earth. National Council for Science and the Environment. Washington DC
- ↑ Caetano-Anolles G, Kim HS, Mittenthal JE (2007). "The origin of modern metabolic networks inferred from phylogenomic analysis of protein architecture". Proc Natl Acad Sci USA 104 (22): 9358–63. Bibcode:2007PNAS..104.9358C. PMC 1890499. PMID 17517598. doi:10.1073/pnas.0701214104.
- ↑ Schmidt S, Sunyaev S, Bork P, Dandekar T (2003). "Metabolites: a helping hand for pathway evolution?". Trends Biochem Sci 28 (6): 336–41. PMID 12826406. doi:10.1016/S0968-0004(03)00114-2.
- ↑ Light S, Kraulis P (2004). "Network analysis of metabolic enzyme evolution in Escherichia coli". BMC Bioinformatics 5: 15. PMC 394313. PMID 15113413. doi:10.1186/1471-2105-5-15. Alves R, Chaleil R, Sternberg M (2002). "Evolution of enzymes in metabolism: a network perspective". J Mol Biol 320 (4): 751–70. PMID 12095253. doi:10.1016/S0022-2836(02)00546-6.
- ↑ Kim HS, Mittenthal JE, Caetano-Anolles G (2006). "MANET: tracing evolution of protein architecture in metabolic networks". BMC Bioinformatics 7: 351. PMC 1559654. PMID 16854231. doi:10.1186/1471-2105-7-351.
- ↑ Teichmann SA, Rison SC, Thornton JM, Riley M, Gough J, Chothia C (2001). "Small-molecule metabolsim: an enzyme mosaic". Trends Biotechnol 19 (12): 482–6. PMID 11711174. doi:10.1016/S0167-7799(01)01813-3.
- ↑ Spirin V, Gelfand M, Mironov A, Mirny L (2006). "A metabolic network in the evolutionary context: Multiscale structure and modularity". Proc Natl Acad Sci USA 103 (23): 8774–9. Bibcode:2006PNAS..103.8774S. PMC 1482654. PMID 16731630. doi:10.1073/pnas.0510258103. Arquivado dende o orixinal o 12 de setembro de 2019. Consultado o 16 de xullo de 2013.
- ↑ Lawrence J (2005). "Common themes in the genome strategies of pathogens". Curr Opin Genet Dev 15 (6): 584–8. PMID 16188434. doi:10.1016/j.gde.2005.09.007. Wernegreen J (2005). "For better or worse: genomic consequences of intracellular mutualism and parasitism". Curr Opin Genet Dev 15 (6): 572–83. PMID 16230003. doi:10.1016/j.gde.2005.09.013.
- ↑ Pál C, Papp B, Lercher M, Csermely P, Oliver S, Hurst L (2006). "Chance and necessity in the evolution of minimal metabolic networks". Nature 440 (7084): 667–70. Bibcode:2006Natur.440..667P. PMID 16572170. doi:10.1038/nature04568.
- ↑ "Metabolism". The Online Etymology Dictionary. Consultado o 2007-02-20.
- ↑ Eknoyan G (1999). "Santorio Sanctorius (1561–1636) – founding father of metabolic balance studies". Am J Nephrol 19 (2): 226–33. PMID 10213823. doi:10.1159/000013455.
- ↑ Williams, H. S. (1904) A History of Science: in Five Volumes. Volume IV: Modern Development of the Chemical and Biological Sciences Harper and Brothers (New York) Retrieved on 2007-03-26
- ↑ Dubos J. (1951). "Louis Pasteur: Free Lance of Science, Gollancz. Quoted in Manchester K. L. (1995) Louis Pasteur (1822–1895)—chance and the prepared mind". Trends Biotechnol 13 (12): 511–515. PMID 8595136. doi:10.1016/S0167-7799(00)89014-9.
- ↑ Kinne-Saffran E, Kinne R (1999). "Vitalism and synthesis of urea. From Friedrich Wöhler to Hans A. Krebs". Am J Nephrol 19 (2): 290–4. PMID 10213830. doi:10.1159/000013463.
- ↑ Eduard Buchner's 1907 Nobel lecture at http://nobelprize.org Accessed 2007-03-20
- ↑ Kornberg H (2000). "Krebs and his trinity of cycles". Nat Rev Mol Cell Biol 1 (3): 225–8. PMID 11252898. doi:10.1038/35043073.
- ↑ Krebs HA, Henseleit K (1932). "Untersuchungen über die Harnstoffbildung im tierkorper". Z. Physiol. Chem. 210: 33–66. doi:10.1515/bchm2.1932.210.1-2.33.
Krebs H, Johnson W (1937). "Metabolism of ketonic acids in animal tissues". Biochem J 31 (4): 645–60. PMC 1266984. PMID 16746382.
Véxase tamén
[editar | editar a fonte]| Vexa a entrada do Galizionario acerca de Metabolismo |
Bibliografía
[editar | editar a fonte]- Rose S, Mileusnic R. The Chemistry of Life. (Penguin Press Science, 1999), ISBN 0-14-027273-9
- Schneider ED, Sagan D. Into the Cool: Energy Flow, Thermodynamics, and Life. (University Of Chicago Press, 2005), ISBN 0-226-73936-8
- Lane N. Oxygen: The Molecule that Made the World. (Oxford University Press, USA, 2004), ISBN 0-19-860783-0
- Price N, Stevens L. Fundamentals of Enzymology: Cell and Molecular Biology of Catalytic Proteins. (Oxford University Press, 1999), ISBN 0-19-850229-X
- Berg J, Tymoczko J, Stryer L. Biochemistry. (W. H. Freeman and Company, 2002), ISBN 0-7167-4955-6
- Cox M, Nelson DL. Lehninger Principles of Biochemistry. (Palgrave Macmillan, 2004), ISBN 0-7167-4339-6
- Brock TD, Madigan MT, Martinko J, Parker J. Brock's Biology of Microorganisms. (Benjamin Cummings, 2002), ISBN 0-13-066271-2
- Da Silva JJRF, Williams RJP. The Biological Chemistry of the Elements: The Inorganic Chemistry of Life. (Clarendon Press, 1991), ISBN 0-19-855598-9
- Nicholls DG, Ferguson SJ. Bioenergetics. (Academic Press Inc., 2002), ISBN 0-12-518121-3
Outros artigos
[editar | editar a fonte]- Respiración celular
- Respiración anaeróbica
- Síntese de proteínas
- Metabolismo basal
- Metabolismo microbiano
- Metabolito secundario
- Clasificación nutricional básica
Ligazóns externas
[editar | editar a fonte]- "Metabolismo". MedlinePlus (en castelán).
- Monografías de metabolismo
- Glosario de térmos bioquímicos
- Glosario de termos biolóxicos e toxicolóxicos Arquivado 20 de setembro de 2007 en Wayback Machine.
- Metabolismo humano
- Dióxido de carbono e metabolismo humano
- Interactive Flow Chart of the Major Metabolic Pathways – Táboa interactiva das rutas metabólicas principais
- Metabolism, Cellular Respiration and Photosynthesis – Metabolismo, respiración celular e fotosíntese
- The Biochemistry of Metabolism – A Bioquímica do Metabolismo
- Topics in Medical Biochemistry – Temas en Bioquímica Médica
- The Medical Biochemistry Page Arquivado 30 de decembro de 2010 en Wayback Machine. – Bioquímica médica
