Proteína periférica de membrana
As proteínas periféricas de membrana son proteínas que se adhiren só temporalmente ás membranas celulares coas cales están asociadas. Estas moléculas están unidas a proteínas integrais de membrana ou penetran nas rexións periféricas da bicapa lipídica da membrana. As subunidades de proteínas regulatorias de moitos canles iónicas e receptores transmembrana, por exemplo, poden definirse como proteínas periféricas de membrana. A diferenza das proteínas integrais de membrana, as periféricas adoitan recollerse no compoñente ou fracción hidrosoluble de todas as proteínas extraídas durante o procedemento de purificación de proteínas. As proteínas cunha áncora GPI son unha excepción a esta regra e poden ter propiedades de purificación similares ás das proteínas integrais de membrana.
A adhesión reversible de proteínas ás membranas biolóxicas pode servir para regular a sinalización celular e moitos outros eventos celulares importantes, por medio de diversos mecanismos.[1] Por exemplo, a estreita asociación entre moitos encimas e membranas celulares pode levalos a unha estreita proximidade cos seus substratos lipídicos.[2] A unión a membranas pode tamén promover o rearranxo, disociación ou cambio conformacional en moitos dominios estruturais de proteínas, orixinando cambios no seu estado de activación e actividade biolóxica.[3][4] Ademais, moitas proteínas están localizadas especificamente na superficie da capa citosólica ou na da capa externa da membrana.[5] Isto facilita a ensamblaxe de complexos multiproteicos ao incrementarse a probabilidade de que se produzan as interaccións proteína-proteína apropiadas.

Unión das proteínas periféricas á bicapa lipídica
[editar | editar a fonte]
As proteínas periféricas de membrana poden interaccionar con outras proteínas ou directamente coa bicapa lipídica. Nese último caso, denomínanse proteínas anfitrópicas.[3] Algunhas proteínas, como as proteínas G e certas proteína quinases, interaccionan simultaneamente coas proteínas transmembrana e a bicapa lipídica. Outras únense primeiro á bicapa lipídica e despois localizan e interaccionan cos seus receptores de membrana.
A bicapa fosfolipídica que forma a superficie das membranas celulares consta dunha rexión central interna hidrofóbica que queda en medio de dúas rexións hidrofílicas, unha na superficie interna e outra na externa da membrana. As superficies interna e externa ou rexións interface do modelo das bicapas de fosfolípidos teñen un grosor de 8 a 10 Å, aínda que pode ser maior en membranas biolóxicas que conteñen unha gran cantidade de gangliósidos ou lipopolisacáridos.[6] A rexión central interna hidrofóbica dunha membrana típica pode ter un grosor de 27 a 32 Å, estimado por dispersón de raios X a baixo ángulo (SAXS, Small angle X-ray scattering).[7] A rexión límite entre a parte interna central hidrofóbica e as rexións interfaciais hidrofílicas é moi estreito, de arredor de 3 Å. A concentración efectiva de auga cambia rapidamente a través desta capa límite ao moverse cara a fóra desde a rexión central hidrofóbica, variando desde case cero a unha concentración de aredor de 2M.[8][9] Os grupos fosfato nas bicapas de fosfolípidos están completamente hidratados ou saturados de auga e están situados a uns 5 Å fóra do límite da rexión central hidrofóbica.[10]
Algunhas proteínas asócianse coas bicapas lipídicas irreversiblemente e poden formar canles transmembrana de hélices alfa ou barril beta. Estas transformacións ocorren en toxinas formadoras de poros como a colicina A, alfa-hemolisina, e outras proteínas. Estas proteínas son descritas xeralmente como periféricas xa que un dos seus estados conformacionais é hidrosoluble ou están só feblemente asociadas coa membrana.[11]
Mecanismos de unión ás membranas
[editar | editar a fonte]
A asociación dunha proteína coa bicapa lipídica pode implicar cambios significativos na estrutura terciaria dunha proteína. Isto pode incluír o pregamento de rexións da estrutura da proteína que estaban previamente sen pregar ou un rearranxo no pregamento ou repregamento da parte da proteína asociada á membrana. Tamén pode implicar a formación ou disociación de estruturas cuaternarias ou complexos oligoméricos, e a unión específica de ións, ligandos, ou lípidos regulatorios.
As proteínas anfitrópicas típicas deben interaccionar fortemente coa bicapa lipídica para realizaren as súas funcións biolóxicas. Estas inclúen o procesamento encimático de lípidos e outras substancias hidrofóbicas, ancoraxe na membrana, e unión e transferencia de pequenos compostos non polares entre diferentes membranas celulares. Estas proteínas poden estar ancoradas á bicapa como resultado de interaccións hidrofóbicas entre a bicapa e residuos non polares expostos na superficie da proteína por interaccións de unión covalente específicas con lípidos regulatorios, ou pola súa unión a áncoras lipídicas unidas covalentemente.
As afinidades de unión á membrana de moitas proteínas periféricas dependen da composición lipídica específica da membrana coa cal están asociadas.[12]
Asociación hidrofóbica non específica
[editar | editar a fonte]As proteínas anfitrópicas asócianse coas bicapas lipídicas por medio de estruturas áncora hidrofóbicas. Ditas hélices α anfifílicas, bucles non polares expostos, residuos de aminoácidos acilados ou lipidados postraducionalmente únense especificamente a lípidos regulatorios como o fosfatidilinositol fosfato.[13]
Áncoras lipídicas unidas covalentemente
[editar | editar a fonte]As proteínas ancoradas a lípidos están unidas covalentemente a cadeas acilo de ácidos graxos no lado citoplasmático da membrana celular por palmitoilación, miristoilación, ou prenilación. Na superfcie da célula, no lado oposto da membrana celular hai proteíans ancoradas a lípidos enlazadas covalentemente aos lípidos glicosilfosfatidilinositol (GPI) e colesterol.[14][15] A asociación de proteínas coas membranas por medio de residuos acilados é un proceso reversible, xa que a cadea acilo pode estar enterrada no peto de unión hidrofóbico da proteína despois da disociación da membrana. Este proceso ocorre nas subunidades beta das proteínas G. Quizais debido a esta necesidade adicional de flexibilidade estrutural, as áncoras lipídicas están xeralmente unidas aos segmentos moi flexibles das estruturas terciarias de proteínas que non son ben resoltos polos métodos de estudo cristalográfico das proteínas.
Unión específica proteína–lípido
[editar | editar a fonte]
Algunhas proteínas citosólicas son recrutadas en diferentes membranas celulares por medio do recoñecemento de certos tipos de lípidos que se encontran en determinadas membranas.[16] A unión dunha proteína a un lípido específico ten lugar por medio de dominios estruturais para a unión á membrana que ten a proteína e que teñen petos de unión específicos para as cabezas polares dos lípidos aos que se unen. Esta é unha típica interacción proteína-ligando, que se estabiliza pola formación de enlaces de hidróxeno intermoleculares, interaccións de van der Waals, e interaccións hidrofóbicas entre as proteína e o ligando lipídico. Tales complexos tamén se estabilizan pola formación de pontes iónicas entre residuos aspartato ou glutamato da proteína e os fosfatos do lípido por medio de ións calcio intercalados (Ca2+). Estas pontes iónicas poden orixinarse e ser estables cando os ións como o Ca2+ están xa unidos a unha proteína en solución, antes de que se una o lípido. A formación de pontes iónicas dáse na interacción proteína-lípido en proteínas con dominio C2 e anexinas.
Interaccións electrostáticas proteína-lípido
[editar | editar a fonte]As proteínas cargadas positivamente son atraídas polas membranas cargadas negativamente por interaccións electrostáticas non específicas. Porén, non todos os péptidos periféricos e proteínas son catiónicos, e só un lado da membrana está cargado negativamente, como ocorre co lado citoplasmático da membrana plasmática, a capa externa da membrana externa das bacterias gramnegativas e das membranas mitocondriais. Por tanto, as interaccións electrostáticas xogan un importante papel na destinación ás membranas de transportadores de electróns como o citocromo c, toxinas catiónicas como a caribdotoxina, e dominios específicos de unión a membrana como algúns dominios PH, dominios C1, e C2.
As interaccións electrostáticas son moi dependentes da forza iónica da solución. Estas interaccións son relativamente febles á forza iónica fisiolóxica (0,14M NaCl): ~3 a 4 kcal/mol para pequenas proteína catiónicas, como o citocromo c, caribdotoxina ou hisactofilina.[13][17][18]
Posición espacial na membrana
[editar | editar a fonte]As orientacións e a profundidade de penetración na membrana de moitas proteínas anfitrópicas e péptidos estudouse usando etiquetado de spin dirixido ao sitio (SDSL, que usa a resonancia do spin do electrón),[19] etiquetado químico, medida das afinidades de unión á membrana de mutantes da proteína,[20] espectroscopia de fluorescencia,[21] espectroscopia NMR en estado sólido ou solución,[22] espectroscopia FTIR ATR,[23] raios X ou difracción de neutróns,[24] e métodos informáticos.[25][26][27][28]
Identificáronse dous modos de asociación das proteínas coa membrana. As proteínas hidrosolubles típicas non teñen residuos non polares expostos nin outras áncoras hidrofóbicas. Por tanto, permanecen completamente en solución acuosa e non penetran na bicapa lipídica, o cal seríalles custoso enerxeticamente. Esas proteínas só interaccionan electrostaticamente coas bicapas; por exemplo, a ribonuclease e a poli-lisina interaccionan coas membranas deste xeito. Porén, as proteínas anfitrópicas típicas teñen varias áncoras hidrofóbicas que penetran a rexión interfacial e chegan á rexión interior hidrocarbonada da membrana. Estas proteínas "deforman" a bicapa lipídica, facendo decrecer a temperatura de transición lípido fluído-xel.[29] A unión é xeralmente unha reacción fortemente exotérmica.[30] A asociación de hélices α anfifílicas coas membranas ocorre de xeito similar.[24][31] As proteínas intrinsicamente desestruturadas ou os péptidos despregados con residuos non polares ou áncoras lipídicas poden tamén penetrar a rexión interfacial da membrana e chegar á parte central hidrocarbonada, especialmente cando tales péptidos son catiónicos e interaccionan con membranas cargadas negativamente.[32][33][34]
Clases de proteínas periféricas
[editar | editar a fonte]Encimas
[editar | editar a fonte]Os encimas periféricos participan no metabolismo de diferentes compoñentes da membrana, como lípidos (fosfolipases e colesterol oxidases), oligosacáridos da parede celular (glicosiltransferases e transglicosidases), ou proteínas (peptidase de sinal e palmitoíl proteín tioesterases). As lipases poden tamén dixerir lípidos que forman micelas ou gotas non polares na auga.
| Clase | Función | Fisioloxía | Estrutura |
|---|---|---|---|
| Pregamento de alfa/beta hidrolase | Cataliza a hidrólise de enlaces químicos.[35] | Inclúen as lipases bacteriana, fúnxica, gástrica e pancreática, as palmitoíl proteín tioesterases, a cutinase, e as colinesterases | [1] |
| Fosfolipase A2 (secretora e citosólica) | Hidrólise do enlace sn-2 dos ácidos graxos de fosfolípidos.[36] | Dixestión lipídica, rotura das membranas e sinalización por lípidos. | [2] [3] |
| Fosfolipase C | Hidroliza o fosfatidilinositol PIP2 formando dous segundos mensaxeiros, inositol trifosfato e diacilglicerol.[37] | Sinalización por lípidos | [4] |
| Colesterol oxidases | Oxidan e isomerizan o colesterol a colest-4-en-3-ona.[38] | Fai diminuír o colesterol da membrana; utilizado na patoxénese bacteriana. | [5] |
| Carotenoide oxixenase | Cliva carotenoides.[39] | Os carotenoides funcionan en plantas e animais a modo de hormonas (incluída a vitamina A en humanos), pigmentos, aromas, recendos florais e compostos de defensa. | [6] |
| Lipoxixenases | Encimas que conteñen ferro que catalizan a dioxixenación de ácidos graxos poliinsaturados.[40] | En animais as lipoxixenases están implicadas na síntese de mediadores inflamatorios chamados leucotrienos. | [7] |
| Alfa toxinas | Clivan fosfolípidos da membrana celular, de modo similar á fosfolipase C.[41] | Patoxénese bacteriana, especialmente de Clostridium perfringens. | [8] |
| Esfingomielinase C | Unha fosfodiesterase que cliva enlaces fosfodiéster.[42] | Procesamento de lípidos como a esfingomielina. | [9] |
| Glicosiltransferases: MurG e transglicosidases | Catalizan a transferencia de restos de azucres desde moléculas doantes activadas a moléculas aceptoras específicas, formando enlaces glicosídicos.[43] | Biosíntese de disacáridos, oligosacáridos e polisacáridos (glicoconxugados); MurG está implicada na biosíntese do peptidoglicano bacteriano. | [10] [11] |
| Ferroquelatase | Converte a protoporfirina IX en hemo.[44] | Implicada no metabolismo da porfirina; as protoporfirinas utilízanse para fortalecer as cascas de ovos. | [12] |
| Familia de proteínas relacionadas coa miotubularina | Lípido fosfatase que desfosforila o fosfatidilinositol 3-fosfato (PtdIns3P) e o fosfatidilinositol (3,5)-bisfosfato (PtdIns(3,5)P2).[45] | Necesarias para a diferenciación da célula muscular. | [13] |
| Dihidroorotato deshidroxenases | Oxidación do dihidroorotato (DHO) a orotato.[46] | Biosíntese de nucleótidos de pirimidina en células procariotas e eucariotas. | [14] |
| Glicolato oxidase | Cataliza a oxidación de α-hidroxiácidos aos correspondentes α-cetoácidos.[47] | En plantas, o encima participa na fotorrespiración. En animais, o encima participa na produción de oxalato. | [15] |
Dominios de destinación a membrana (“grampas lipídicas”)
[editar | editar a fonte]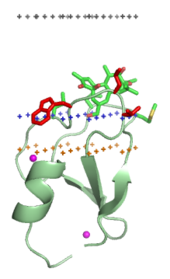
Os dominios que marcan o destino dunha proteína como proteína de membrana están asociados especificalmente coas cabezas polares dos seus ligandos lipídicos integrados nas membranas. Estes ligandos lipídicos están presentes en distintas concentracións nos diferentes tipos de membranas celulares (por exemplo, o PtdIns3P atópase na maioría das membranas dos endosomas temperáns, o PtdIns(3,5)P2 na dos endosomas tardíos, e o PtdIns4P nas do aparato de Golgi).[16] Por tanto, cada dominio destina a unha proteína a unha membrana específica. Estes dominios son:
- Dominio C1 [16], que se une ao diacilglicerol e aos ésteres do forbol.
- Dominio C2 [17], que se une á fosfatidilserina ou á fosfatidilcolina.
- Dominio de homoloxía de pleckstrina [18], dominio PX [19], e dominio Tubby [20], que se unen a diferentes fosfoinosítidos.
- Dominio FYVE [21], que son máis específicos para o PtdIns3P.
- Dominio ENTH [22], que se une ao PtdIns(3,4)P2 ou ao PtdIns(4,5)P2.
- Dominio ANTH [23], que se une ao PtdIns(4,5)P2.
- Proteínas da familia ERM (ezrina/radixina/moesina) [24], que se unen ao PtdIns(4,5)P2.
- Outros dominios de proteínas que se unen a fosfoinosítidos son o dominio de unión á fosfotirosina [25] e certos dominio PDZ. Únense ao PtdIns(4,5)P2.
- Dominios discoidina de factores de coagulación do sangue. [26].
- Dominios ENTH, VHS e ANTH [27].
Dominios estruturais
[editar | editar a fonte]Os dominios estruturais median a unión doutras proteínas á membrana. Esta unión a membranas pode ser mediada por ións calcio (Ca2+) que forman pontes entre os residuos ácidos da proteína e grupos fosfato de lípidos, como ocorre nas anexinas ou no dominio GLA.
| Clase | Función | Fisioloxía | Estrutura |
|---|---|---|---|
| As anexinas | Unión a membrana intracelular/fosfolípido dependente do calcio.[48] | Entre as funcións están o tráfico de vesículas, fusión de membranas e formación de canles iónicas. | [28] |
| Sinapsina I | Cobre as vesículas sinápticas e únese a varios elementos do citoesqueleto.[49] | Funcións na regulación de liberación de neurotransmisores. | [29] |
| Sinucleína | Funcións celulares descoñecidas.[50] | Crese que xoga un papel na regulación da estabilidade e renovación da membrana plasmática. Asociado coa enfermidade de Parkinson e a de Alzheimer. | [30] |
| Dominios GLA do sistema de coagulación do sangue | Os dominios gamma-carboxiglutamato (GLA) son responsables da grande afinidade de unión aos ións calcio.[51] | Implicados na función de factores de coagulación na fervenza de coagulación do sangue. | [31] |
| Espectrina e α-actinina-2 | Atópanse en varias proteínas do citoesqueleto e microfilamentos.[52] | Mantemento da integridade da membrana plasmática e estrutura do citoesqueleto. | [32] |
Transportadores de pequenas moléculas hidrofóbicas
[editar | editar a fonte]Estas proteínas periféricas funcionan como transportadores de compostos non polares entre diferentes tipos de membranas celulares ou entre as membranas e os complexos proteicos citosólicos. As substancias transportadas son o fosfatidilinositol, tocoferol, gangliósidos, glicolípidos, derivados de esterois, retinol, ácidos graxos, auga, macromoléculas, glóbulos vermellos do sangue, fosfolípidos, e nucleótidos. Entre estas proteínas están:
- Proteína de transferencia de glicolípidos [33]
- Lipocalinas, entre as que están a proteína de unión ao retinol e proteínas de unión a ácidos graxos [34]
- Proteínas de unión a poliisoprenoides [35]
- Proteína activadora do gangliósido GM2 [36]
- Dominio CRAL-TRIO (proteínas de transferencia de α-tocoferol e fosfatidilinositol sec14p) [37]
- Proteína transportadora de esterol [38]
- Proteínas de transferencia de fosfatidilinositol e dominios STAR [39]
- Proteína de unión ao oxisterol [40]
Transportadores de electróns
[editar | editar a fonte]Estas proteínas están implicadas na cadea de transporte de electróns. Entre elas están o citocromo c, cupredoxinas, proteínas ferrosulfuradas de alto potencial, adrenodoxina redutase, algunhas flavoproteínas, e outras.
Hormonas polipeptídicas, toxinas, e péptidos antimicrobianos
[editar | editar a fonte]Moitas hormonas, toxinas, inhibidores, ou péptidos antimicrobianos interaccionan especificamente con complexos de proteínas transmembrana. Poden tamén acumularse na superficie da bicapa lipídica antes de unirse ás súas proteínas diana. Ditos ligandos polipéptidos están con frecuencia cargados positivamente e interaccionan electrostaticamente con membranas aniónicas.
Algunhas proteínas hidrosolubles e péptidos poden tamén formar canles transmembrana. Xeralmente sofren oligomerización, cambios conformacionais significativos, e asócianse coas membranas irreversiblemente. Determinouse a estrutura tridimensional dun destas canles transmembrana, o da α-hemolisina. Noutros casos, a estrutura experimental representa unha conformación hidrosoluble que interacciona coa bicapa lipídica perifericamente, aínda que algúns dos péptidos que forman canles son bastante hidrofóbicos e, xa que logo, foron estudados por espectroscopía NMR en solventes orgánicos ou en presenza de micelas.
| Clase | Proteínas | Fisioloxía |
|---|---|---|
| Toxinas de velenos |
|
Tipos ben coñecidos de biotoxinas son as neurotoxinas, citotoxinas, hemotoxinas e necrotoxinas. As biotoxinas teñen dúas funcións principais: predación (toxinas de serpe, escorpión e caracol mariño Conus) e defensa (toxinas da abella e de formiga).[53] |
| Toxinas de anemone |
|
A inhibición de canles de sodio e potasio e formación de poros de membrana son as principais accións dunhas 40 toxinas peptídicas coñecidas de anemones. As anemones de mar son animais carnívoros e usan toxinas na súa predación e defensa; a toxina das anemones ten unha toxicidade similar á dos organofosfatos máis tóxicos usados como axentes da guerra química.[54] |
| Toxinas bacterianas |
|
As toxinas microbianas son os principais factores de virulencia para diversas bacterias patóxenas. Algunhas toxinas, son toxinas formadoras de poros que lisan as membranas celulares. Outras toxinas inhiben a biosíntese de proteínas ou activan as vías dos segundos mensaxeiros causando grandes alteracións nas vías de transdución de sinais fundamentais para o mantemento de diversas funcións celulares. Varias toxinas bacterianas poden actuar directamente sobre o sistema inmunitario, ao funcionaren como superantíxenos e causaren unha masiva proliferación de células T, que sobreactiva o sistema inmunitario. A toxina botulínica é unha neurotoxina que impide que vesículas neurosecretoras se unan ou fusionen coa membrana plasmática sináptica do nervio, inhibindo a liberación de neurotransmisores.[55] |
| Toxinas fúnxicas |
|
Estes péptidos están caracterizados pola presenza dun aminoácido pouco usual, o ácido α-aminoisobutírico, e presentan propiedades antibióticas e antifúnxicas debido ás súas actividades de formación de canles nas membranas.[56] |
| Péptidos antimicrobianos | Os modos de acción polo scales os péptidos antimicrobianos matan as bacterias é variado e inclúen a rotura das membranas, a interferencia co metabolismo, e afectando a compoñentes citoplásmicos. A diferenza de moitos antibióticos convencionais estes péptidos parecen ser bactericidas en vez de bacteriostáticos. | |
| Defensinas | As defensinas son un tipo de péptido antimicrobiano; e son un importante compoñente de virtualmente todas as defensas inmunitarias innatas contra as invasións microbianas. As defensinas penetran nas membranas das células microbianas por medio de atraccións eléctricas, e forman un poro na membrana permitindo o fluxo, que acaba producindo a lise dos microorganismos.[57] | |
| Péptidos neuronais |
|
Estas proteínas excitan as neuronas, producen respostas de comportamento, son potentes vasodilatadores, e son responsables da contracción en moitos tipos de músculos lisos.[58] |
| Reguladores da apoptose | Membros da familia Bcl-2 controlan a permeabilidade da membrana mitocondrial externa. O propio Bcl-2 suprime a apoptose en varios tipos de células como os linfocitos e neuronas. |
Notas
[editar | editar a fonte]- ↑ David S. Cafiso Structure and interactions of C2 domains at membrane surfaces. In: Tamm LK (Editor) (2005). Protein-Lipid Interactions: From Membrane Domains to Cellular Networks. Chichester: John Wiley & Sons. pp. 403–22. ISBN 3-527-31151-3.
- ↑ Ghosh M; Tucker; DE.; et al. (2006). "Properties of group IV phospholipase A2 family (review)". Prog. Lipid. Res. 45 (6): 487–510. PMID 16814865. doi:10.1016/j.plipres.2006.05.003.
- ↑ 3,0 3,1 Johnson J, Cornell R (2002). "Amphitropic proteins: regulation by reversible membrane interactions (review)". Mol Membr Biol 16 (3): 217–35. PMID 10503244. doi:10.1080/096876899294544.
- ↑ Guruvasuthevan RT, Craig JW; et al. (2006). "Evidence that membrane insertion of the cytosolic domain of Bcl-xL is governed by an electrostatic turtle suny mechanism". J. Mol. Biol. 359 (4): 1045–1058. PMID 16650855. doi:10.1016/j.jmb.2006.03.052.
- ↑ Takida S and Wedegaertner PB (2004). "Exocytic pathway-independent plasma membrane targeting of heterotrimeric G proteins". FEBS Letters 567 (2–3): 209–213. PMID 15178324. doi:10.1016/j.febslet.2004.04.062.
- ↑ McIntosh, TJ; Vidal A, Simon SA (2003). The energetics of peptide-lipid interactions: modification by interfacial dipoles and cholesterol. In Current Topics in Membranes (52). Academic Press. pp. 205–253. ISBN 978-0-12-643871-0.
- ↑ Mitra K, Ubarretxena-Belandia I, Taguchi T, Warren G, Engelman D (2004). "Modulation of the bilayer thickness of exocytic pathway membranes by membrane proteins rather than cholesterol". Proc Natl Acad Sci USA 101 (12): 4083–4088. Bibcode:2004PNAS..101.4083M. PMC 384699. PMID 15016920. doi:10.1073/pnas.0307332101.
- ↑ Marsh D (2001). "Polarity and permeation profiles in lipid membranes". Proc Natl Acad Sci USA 98 (14): 7777–7782. Bibcode:2001PNAS...98.7777M. PMC 35418. PMID 11438731. doi:10.1073/pnas.131023798.
- ↑ Marsh D (2002). "Membrane water-penetration profiles from spin labels". Eur Biophys J 31 (7): 559–562. PMID 12602343. doi:10.1007/s00249-002-0245-z.
- ↑ Nagle J, Tristram-Nagle S (2000). "Structure of lipid bilayers". Biochim Biophys Acta 1469 (3): 159–195. PMC 2747654. PMID 11063882. doi:10.1016/S0304-4157(00)00016-2.
- ↑ Goñi F (2002). "Non-permanent proteins in membranes: when proteins come as visitors (Review)". Mol Membr Biol 19 (4): 237–45. PMID 12512770. doi:10.1080/0968768021000035078.
- ↑ McIntosh T, Simon S (2006). "Roles of bilayer material properties in function and distribution of membrane proteins". Annu Rev Biophys Biomol Struct 35 (1): 177–198. PMID 16689633. doi:10.1146/annurev.biophys.35.040405.102022. Arquivado dende o orixinal o 15 de setembro de 2019. Consultado o 04 de xaneiro de 2014.
- ↑ 13,0 13,1 Hanakam F, Gerisch G, Lotz S, Alt T, Seelig A (1996). "Binding of hisactophilin I and II to lipid membranes is controlled by a pH-dependent myristoyl-histidine switch". Biochemistry 35 (34): 11036–11044. PMID 8780505. doi:10.1021/bi960789j.
- ↑ Silvius, JR (2003). Lipidated peptides as tools for understanding the membrane interactions of lipid-modified proteins. In Current Topics in Membranes (52). Academic Press. pp. 371–395. ISBN 978-0-12-643871-0.
- ↑ Baumann, NA; Mennon AK (2002). Lipid modifications of proteins. In DE Vance and JE Vance (Eds.) Biochemistry of Lipids, Lipoproteins and Membranes (4th ed.). Elsevier Science. pp. 37–54. ISBN 978-0-444-51139-3.
- ↑ 16,0 16,1 Cho, W. and Stahelin, R.V. (2005). "Membrane-protein interactions in cell signaling and membrane trafficking". Annual Review of Biophysics and Biomolecular Structure 34: 119–151. PMID 15869386. doi:10.1146/annurev.biophys.33.110502.133337. Arquivado dende o orixinal o 15 de setembro de 2019. Consultado o 2007-01-23.
- ↑ Ben-Tal N, Honig B, Miller C, McLaughlin S. (1997). "Electrostatic binding of proteins to membranes. Theoretical predictions and experimental results with charybdotoxin and phospholipid vesicles". Biophys J. 73 (4): 1717–1727. Bibcode:1997BpJ....73.1717B. PMC 1181073. PMID 9336168. doi:10.1016/S0006-3495(97)78203-1.
- ↑ Sankaram, MB; Marsh D (1993). Protein-lipid interactions with peripheral membrane proteins. In: Protein-lipid interactions (Ed. A. Watts). Elsevier. pp. 127–162. ISBN 0-444-81575-9.
- ↑ Malmberg N, Falke J (2005). "Use of EPR power saturation to analyze the membrane-docking geometries of peripheral proteins: applications to C2 domains". Annu Rev Biophys Biomol Struct 34 (1): 71–90. PMID 15869384. doi:10.1146/annurev.biophys.34.040204.144534. Arquivado dende o orixinal o 15 de setembro de 2019. Consultado o 04 de xaneiro de 2014.
- ↑ Spencer A, Thuresson E, Otto J, Song I, Smith T, DeWitt D, Garavito R, Smith W (1999). "The membrane binding domains of prostaglandin endoperoxide H synthases 1 and 2. Peptide mapping and mutational analysis". J Biol Chem 274 (46): 32936–32942. PMID 10551860. doi:10.1074/jbc.274.46.32936.
- ↑ Lathrop B, Gadd M, Biltonen R, Rule G (2001). "Changes in Ca2+ affinity upon activation of Agkistrodon piscivorus piscivorus phospholipase A2". Biochemistry 40 (11): 3264–3272. PMID 11258945. doi:10.1021/bi001901n.
- ↑ Kutateladze T, Overduin M (2001). "Structural mechanism of endosome docking by the FYVE domain". Science 291 (5509): 1793–1796. Bibcode:2001Sci...291.1793K. PMID 11230696. doi:10.1126/science.291.5509.1793.
- ↑ Tatulian S, Qin S, Pande A, He X (2005). "Positioning membrane proteins by novel protein engineering and biophysical approaches". J Mol Biol 351 (5): 939–947. PMID 16055150. doi:10.1016/j.jmb.2005.06.080.
- ↑ 24,0 24,1 Hristova K, Wimley WC, Mishra VK, Anantharamiah GM, Segrest JP, White SH. (1999). "An amphipathic alpha-helix at a membrane interface: a structural study using a novel X-ray diffraction method". J Mol Biol 290 (1): 99–117. PMID 10388560. doi:10.1006/jmbi.1999.2840.
- ↑ Murray D, Honig B (2002). "Electrostatic control of the membrane targeting of C2 domains". Mol Cell 9 (1): 145–154. PMID 11804593. doi:10.1016/S1097-2765(01)00426-9.
- ↑ Efremov R, Nolde D, Konshina A, Syrtcev N, Arseniev A (2004). "Peptides and proteins in membranes: what can we learn via computer simulations?". Curr Med Chem 11 (18): 2421–42. PMID 15379706.
- ↑ Lomize A, Pogozheva I, Lomize M, Mosberg H (2006). "Positioning of proteins in membranes: a computational approach". Protein Sci 15 (6): 1318–1333. PMC 2242528. PMID 16731967. doi:10.1110/ps.062126106.
- ↑ Lomize A, Lomize M, Pogozheva I. "Comparison with experimental data". Orientations of Proteins in Membranes. University of Michigan. Consultado o 2007-02-08.
- ↑ Papahadjopoulos D, Moscarello M, Eylar E, Isac T (1975). "Effects of proteins on thermotropic phase transitions of phospholipid membranes". Biochim Biophys Acta 401 (3): 317–335. PMID 52374. doi:10.1016/0005-2736(75)90233-3.
- ↑ Seelig J (2004). "Thermodynamics of lipid-peptide interactions". Biochim Biophys Acta 1666 (1–2): 40–50. PMID 15519307. doi:10.1016/j.bbamem.2004.08.004.
- ↑ Darkes MJM, Davies SMA, Bradshaw JP (1997). "Interaction of tachykinins with phospholipid membranes: A neutron diffraction study". Physica B 241: 1144–1147. Bibcode:1998PhyB..241.1144D. doi:10.1016/S0921-4526(97)00811-9.
- ↑ Ellena JF, Moulthrop J, Wu J, Rauch M, Jaysinghne S, Castle JD, Cafiso DS. (2004). "Membrane position of a basic aromatic peptide that sequesters phosphatidylinositol 4,5 bisphosphate determined by site-directed spin labeling and high-resolution NMR". Biophys J. 87 (5): 3221–3233. Bibcode:2004BpJ....87.3221E. PMC 1304792. PMID 15315949. doi:10.1529/biophysj.104.046748.
- ↑ Marcotte I, Dufourc E, Ouellet M, Auger M (2003). "Interaction of the neuropeptide met-enkephalin with zwitterionic and negatively charged bicelles as viewed by 31P and 2H solid-state NMR". Biophys J 85 (1): 8105–8109. PMC 1303088. PMID 12829487. doi:10.1021/bi0341859.
- ↑ Zhang W, Crocker E, McLaughlin S, Smith S (2003). "Binding of peptides with basic and aromatic residues to bilayer membranes: phenylalanine in the myristoylated alanine-rich C kinase substrate effector domain penetrates into the hydrophobic core of the bilayer". J Biol Chem 278 (24): 21459–21466. PMID 12670959. doi:10.1074/jbc.M301652200.
- ↑ "Pfam entry Abhydrolase 1". Arquivado dende o orixinal o 29 de setembro de 2007. Consultado o 04 de xaneiro de 2014.
- ↑ "Pfam entry: Phospholipase A2". Arquivado dende o orixinal o 29 de setembro de 2007. Consultado o 04 de xaneiro de 2014.
- ↑ "Pfam entry: Phosphatidylinositol-specific phospholipase C, X domain". Arquivado dende o orixinal o 29 de setembro de 2007. Consultado o 04 de xaneiro de 2014.
- ↑ "Pfam entry: Cholesterol oxidase". Arquivado dende o orixinal o 29 de setembro de 2007. Consultado o 04 de xaneiro de 2014.
- ↑ "Pfam entry: Retinal pigment epithelial membrane protein". Arquivado dende o orixinal o 29 de setembro de 2007. Consultado o 04 de xaneiro de 2014.
- ↑ "Pfam entry: Lipoxygenase". Arquivado dende o orixinal o 29 de setembro de 2007. Consultado o 04 de xaneiro de 2014.
- ↑ PDBsum entry: Alpha Toxin
- ↑ "Pfam entry: Type I phosphodiesterase". Arquivado dende o orixinal o 29 de setembro de 2007. Consultado o 04 de xaneiro de 2014.
- ↑ "Pfam entry: Glycosyl transferases group 1". Arquivado dende o orixinal o 29 de setembro de 2007. Consultado o 04 de xaneiro de 2014.
- ↑ "Pfam entry: Ferrochelatase". Arquivado dende o orixinal o 29 de setembro de 2007. Consultado o 04 de xaneiro de 2014.
- ↑ "Pfam entry:Myotubularin-related". Arquivado dende o orixinal o 26 de setembro de 2007. Consultado o 04 de xaneiro de 2014.
- ↑ "Pfam entry:Dihydroorotate dehydrogenase". Arquivado dende o orixinal o 26 de setembro de 2007. Consultado o 04 de xaneiro de 2014.
- ↑ "Pfam entry: FMN-dependent dehydrogenase". Arquivado dende o orixinal o 29 de setembro de 2007. Consultado o 04 de xaneiro de 2014.
- ↑ "entrada de Pfam da anexina". Arquivado dende o orixinal o 29 de setembro de 2007. Consultado o 04 de xaneiro de 2014.
- ↑ "entrada de Pfam da sinapsina N". Arquivado dende o orixinal o 26 de setembro de 2007. Consultado o 04 de xaneiro de 2014.
- ↑ "entrada de Pfam da sinucleína". Arquivado dende o orixinal o 26 de setembro de 2007. Consultado o 04 de xaneiro de 2014.
- ↑ "entrada de Pfam de Gla". Arquivado dende o orixinal o 29 de setembro de 2007. Consultado o 04 de xaneiro de 2014.
- ↑ "entrada de Pfam da espectrina". Arquivado dende o orixinal o 26 de setembro de 2007. Consultado o 04 de xaneiro de 2014.
- ↑ Herv ̌Rochat, Marie-France Martin-Eauclaire (editors) (2000). Animal toxins: facts and protocols. Basel: Birkhũser Verlag. ISBN 3-7643-6020-8.
- ↑ Patocka, Jiri and Anna Strunecka. (1999) Ver toxina da anemone. Arquivado 15 de xuño de 2013 en Wayback Machine. The ASA Newsletter.
- ↑ Schmitt C, Meysick K, O'Brien A (1999). "Bacterial toxins: friends or foes?". Emerg Infect Dis 5 (2): 224–234. PMC 2640701. PMID 10221874. doi:10.3201/eid0502.990206. Arquivado dende o orixinal o 26 de abril de 2011. Consultado o 04 de xaneiro de 2014.
- ↑ Chugh J, Wallace B (2001). "Peptaibols: models for ion channels" (PDF). Biochem Soc Trans 29 (Pt 4): 565–70. PMID 11498029. doi:10.1042/BST0290565. Arquivado dende o orixinal (PDF) o 26 de xaneiro de 2021. Consultado o 04 de xaneiro de 2014.
- ↑ Oppenheim1, J J, A Biragyn2, L W Kwak2 and D Yang (2003). "Roles of antimicrobial peptides such as defensins in innate and adaptive immunity". Annals of the Rheumatic Diseases 62: ii17–21. PMC 1766745. PMID 14532141.
- ↑ "Pfam entry Tachykinin". Arquivado dende o orixinal o 26 de setembro de 2007. Consultado o 04 de xaneiro de 2014.
Referencias xerais
[editar | editar a fonte]- Lukas K. Tamm (Editor) (2005). Protein-Lipid Interactions: From Membrane Domains to Cellular Networks. Chichester: John Wiley & Sons. ISBN 3-527-31151-3.
- Cho, W. and Stahelin, R.V. (2005). "Membrane-protein interactions in cell signaling and membrane trafficking". Annual Review of Biophysics and Biomolecular Structure 34 (1): 119–151. PMID 15869386. doi:10.1146/annurev.biophys.33.110502.133337. Arquivado dende o orixinal o 15 de setembro de 2019. Consultado o 2007-01-23.
- Goni F.M. (2002). "Non-permanent proteins in membranes: when proteins come as visitors" (PDF). Mol. Membr. Biol 19 (4): 237–245. PMID 12512770. doi:10.1080/0968768021000035078.
- Johnson J, Cornell R (1999). "Amphitropic proteins: regulation by reversible membrane interactions (review)" (PDF). Mol Membr Biol 16 (3): 217–235. PMID 10503244. doi:10.1080/096876899294544.
- Seaton B.A. e Roberts M.F. Peripheral membrane proteins. pp. 355–403. In Biological Membranes (Eds. K. Mertz e B.Roux), Birkhauser Boston, 1996.
- Benga G. Protein-lipid interactions in biological membranes, pp. 159–188. In Structure and Properties of Biological Membranes, vol. 1 (Ed. G. Benga) Boca Raton CRC Press, 1985.
- Kessel A. e Ben-Tal N. 2002. Free energy determinants of peptide association with lipid bilayers. In Current Topics in Membranes 52: 205–253.
- Malmberg N, Falke J (2005). "Use of EPR power saturation to analyze the membrane-docking geometries of peripheral proteins: applications to C2 domains". Annu Rev Biophys Biomol Struct 34 (1): 71–90. PMID 15869384. doi:10.1146/annurev.biophys.34.040204.144534. Arquivado dende o orixinal o 15 de setembro de 2019. Consultado o 04 de xaneiro de 2014.
- McIntosh T, Simon S (2006). "Roles of bilayer material properties in function and distribution of membrane proteins". Annu Rev Biophys Biomol Struct 35 (1): 177–198. PMID 16689633. doi:10.1146/annurev.biophys.35.040405.102022. Arquivado dende o orixinal o 15 de setembro de 2019. Consultado o 04 de xaneiro de 2014.
Véxase tamén
[editar | editar a fonte]Outros artigos
[editar | editar a fonte]- Proteína de membrana
- Lipoproteína
- Proteína transmembrana
- Proteína integral de membrana
- Péptido antimicrobiano
Ligazóns externas
[editar | editar a fonte]- UMichOPM classes, type 2
- DOLOP Base de datos de lipoproteínas bacterianas orientado á xenómica
- Base de datos Peptaibol Arquivado 27 de xaneiro de 2011 en Wayback Machine.
- Base de datos de péptidos antimicrobianosArquivado 20 de xullo de 2011 en Wayback Machine.
